Mycoplasma laboratorium
| Mycoplasma mycoides JCVI-syn1.0 | ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Біологічна класифікація | ||||||||||||||||
| ||||||||||||||||
| Mycoplasma mycoides JCVI-syn1.0 Gibson et al., 2010[a 1] | ||||||||||||||||
| Синоніми[a 2] | ||||||||||||||||
| Mycoplasma laboratorium Reich, 2000 | ||||||||||||||||
Посилання
| ||||||||||||||||
|
Mycoplasma laboratorium або Synthia[b 1] (бактерія Синтія) — штучно синтезований штам бактерії. Проєкт побудови нової бактерії розвивався з моменту її створення. Спочатку метою було виявити мінімальний набір генів, необхідний для підтримки життя з геному Mycoplasma genitalium і синтезувати ці гени синтетично, щоб створити «новий» організм. Mycoplasma genitalium спочатку була обрана як основа для цього проєкту, оскільки на той час у неї була найменша кількість генів з усіх проаналізованих організмів. Пізніше, фокус перемикнувся на Mycoplasma mycoides і застосування підходу «спроб і помилок»[b 2].
Щоб визначити мінімальний набір генів, необхідний для підтримки життя, кожен з 482 генів M. genitalium був індивідуально видалений і життєздатність отриманого мутанта перевірена. Це призвело до визначення мінімального набору з 382 генів, які теоретично повинні представляти мінімальний геном[a 3]. У 2008 році повний набір генів M. genitalium був побудований в лабораторії з додаванням «водяних знаків» для ідентифікації генів як синтетичних[b 3][a 4]. Однак M. genitalium росте надзвичайно повільно і М. mycoides було обрано як нову ціль, щоб прискорити експерименти, спрямовані на визначення набору генів, фактично необхідного для росту[b 4].
У 2010 році повний геном M. mycoides був успішно синтезований з комп'ютерних записів і пересаджений в існуючу клітину Mycoplasma capricolum, в якої було видалено ДНК[b 5]. За підрахунками, синтетичний геном, використаний для цього проєкту, коштував 40 мільйонів доларів США та 200 людино-років[b 4]. Нова бактерія була здатна рости і отримала назву JCVI-syn1.0, або Synthia. Після додаткових експериментів з виявлення меншого набору генів, який міг би продукувати функціональний організм, виготовлено JCVI-syn3.0, що містить 473 гени[b 2]. 149 з цих генів мають невідому функцію[b 2]. Оскільки геном JCVI-syn3.0 є новим, його вважають першим справді синтетичним організмом.
Проєкт мінімального геному[ред. | ред. код]
Виготовлення Synthia є замислом у галузі синтетичної біології, реалізованим в Інституті Дж. Крейга Вентера групою з приблизно 20 вчених на чолі з Нобелівським лауреатом Гемілтоном Смітом, яка містила також дослідника ДНК Крейга Вентера та мікробіолога Клайда А. Гатчісона III. Загальною метою є зведення живого організму до найнеобхіднішого в ньому і, таким чином, з'ясування того, що потрібно для побудови нового організму з нуля[a 3]. Спершу увага була зосереджена на бактерії M. genitalium, облігатному внутрішньоклітинному паразиті, геном якого містить 482 гени, які утворені 582970 пар основ, розташованих в одній кільцевій хромосомі (на момент початку проєкту це був найменший геном серед усіх відомих у природних організмів, які можна вирощувати у культурі вільних клітин). Вони використовували транспозоновий мутагенез, щоб визначити гени, які не були необхідними для росту організму, що призвело до виявлення мінімального набору з 382 генів[a 3]. Цей замисел відомий як проєкт мінімального геному[a 5].
Вибір організму[ред. | ред. код]
Мікоплазма[ред. | ред. код]
Mycoplasma — рід бактерій класу Mollicutes у відділі Tenericutes, що характеризується відсутністю клітинної стінки (що робить її грам-негативною) через паразитичний або коменсальний спосіб життя. У молекулярній біології цьому роду приділяють багато уваги як через те, що він є сумнозвісним важко викорінюваним забруднювачем у культурах клітин ссавців (він несприйнятливий до бета-лактамних та інших антибіотиків)[a 6], так і через можливість потенційного використання його як модельного організму через невеликий розмір його геному[a 7]. Вибір роду для проєкту Synthia датується 2000-м роком, коли Карл Райх придумав фразу Mycoplasma laboratorium[a 2].
Інші організми з малими геномами[ред. | ред. код]
Станом на 2005 рік, Pelagibacter ubique (α-протеобактерія порядку Rickettsiales) мав найменший відомий геном (1 308 759 пар основ) серед будь-яких вільних живих організмів і є однією з найменших відомих самовідтворювальних клітин. Це, мабуть, найчисленніша бактерія у світі (можливо 1028 окремих клітин) і, за оцінками, разом з іншими членами клади SAR11, вони складають від чверті до половини всіх бактеріальних або археальних клітин в океані[a 8]. Вона була ідентифікована у 2002 році за послідовностями рРНК і була повністю секвенована у 2005 році[a 9]. Надзвичайно важко вирощувати види, які не досягають високої щільності росту в лабораторній культурі[a 10][a 11]. Кілька нещодавно відкритих видів мають менше генів, ніж M. genitalium, але не є вільноживучими: багато важливих генів, яких немає у Hodgkinia cicadicola, Sulcia muelleri, Baumannia cicadellinicola (симбіонти цикад) та Carsonella ruddi (симбіонт листоблішки Pachypsylla venusta[a 12]) можуть кодуватися в ядрі хазяїна[a 13]. Організмом з найменшим відомим набором генів станом на 2013 рік була Nasuia deltocephalinicola, облігатний симбіонт. Він має лише 137 генів і розмір геному 112 тисяч пар основ[a 14][b 6].
| видова назва | кількість генів | розмір (т.п.о.) |
|---|---|---|
| Candidatus Hodgkinia cicadicola Dsem [1] [Архівовано 3 липня 2019 у Wayback Machine.] | 169 | 0.14 |
| Candidatus Carsonella ruddii PV [2] [Архівовано 4 липня 2019 у Wayback Machine.] | 182 | 0.16 |
| Candidatus Sulcia muelleri GWSS [3] [Архівовано 3 липня 2019 у Wayback Machine.] | 227 | 0.25 |
| Candidatus Sulcia muelleri SMDSEM [4] [Архівовано 3 липня 2019 у Wayback Machine.] | 242 | 0.28 |
| Buchnera aphidicola str. Cinara cedri [5] [Архівовано 4 липня 2019 у Wayback Machine.] | 357 | 0.4261 |
| Mycoplasma genitalium G37 [6] [Архівовано 6 травня 2021 у Wayback Machine.] | 475 | 0.58 |
| Candidatus Phytoplasma mali [7] [Архівовано 4 липня 2019 у Wayback Machine.] | 479 | 0.6 |
| Buchnera aphidicola str. Baizongia pistaciae [8] [Архівовано 4 липня 2019 у Wayback Machine.] | 504 | 0.6224 |
| Nanoarchaeum equitans Kin4-M [9] [Архівовано 3 липня 2019 у Wayback Machine.] | 540 | 0.49 |
Техніки[ред. | ред. код]
Для проєкту потрібно було розробити або адаптувати кілька лабораторних технік, оскільки він потребував синтез та маніпуляції з дуже великими ділянками ДНК.
Трансплантація бактеріального геному[ред. | ред. код]
У 2007 році команда Вентера повідомила, що їм вдалося перенести хромосому виду Mycoplasma mycoides до Mycoplasma capricolum шляхом:
- виділення геному M. mycoides: м’який лізис клітин, що потрапили в агар - розплавлений агар, змішаний з клітинами та залишений для утворення гелю, з подальшим електрофорезом в пульсувальному полі та виділення смуги правильного розміру (кільцева; 1,25 тисячі пар основ);
- створення компетентних клітин-реципієнтів M. capricolum: ріст у збагачених середовищах супроводжувався голодуванням у збіднених середовищах, де нуклеотидне голодування призводить до інгібування реплікації ДНК та зміни морфології; і
- поліетиленгліколь-опосередкована трансформація кільцевої хромосоми до клітин, що не містять ДНК, з подальшим відбором[a 15].
Термін трансформація використовується для позначення вставлення вектора в бактеріальну клітину (електропорацією або тепловим шоком). Тут, термін трансплантація застосовується як ядерна трансплантація.
Синтез бактеріальної хромосоми[ред. | ред. код]
У 2008 році група Вентера описала виготовлення синтетичного геному, копії послідовності L43967 [Архівовано 5 жовтня 2016 у Wayback Machine.] M. genitalium G37, за ієрархічною стратегією[a 16]:
- Синтез → 1 тисячі пар основ (т.п.о.): Геномна послідовність була синтезована Blue Heron [Архівовано 28 січня 2021 у Wayback Machine.] у 1078 касетах 1080 п.о. із перекриттям 80 п.о. і сайтами рестрикції NotI (неефективна, але рідкісна рестриктаза).
- Лігування → 10 т.п.о.: 109 груп серії з 10 послідовних касет лігували та клонували в E. coli в плазміді й правильну перестановку перевіряли секвенуванням.
- Мультиплексна ПЛР → 100 т.п.о.: 11 груп серії з 10 послідовних 10 т.п.о. збірок (вирощених на дріжджах) об'єднували мультиплексною ПЛР, використовуючи пару праймерів для кожної 10 т.п.о. збірки.
- Виділення та рекомбінація → вторинні вузли були виділені, з’єднані та трансформовані в дріжджові сферопласти без векторної послідовності (присутній у збірці 811-900).
Геном цього результату 2008 року, M. genitalium JCVI-1.0, опублікований на GenBank як CP001621.1. Його не слід плутати з пізнішими синтетичними організмами, позначеними як JCVI-syn, на основі M. mycoides[a 16] .
Синтетичний геном[ред. | ред. код]
У 2010 році Вентер та його колеги створили штам Mycoplasma mycoides JCVI-syn1.0 із синтетичним геномом[a 1]. Спочатку синтетична конструкція не працювала, щоб точно визначити помилку, що призвела до затримки всього проєкту на 3 місяці, була створена серія напівсинтетичних конструкцій[b 4]. Причиною збою був однонуклеотидний зсув рамки зчитування в DnaA, факторі ініціації реплікації[a 1].
Метою побудови клітини з синтетичним геномом було випробування методології як кроку до створення модифікованих геномів у майбутньому. Використання природного геному як шаблону мінімізувало потенційні джерела невдач. Кілька відмінностей є у Mycoplasma mycoides JCVI-syn1.0 щодо референсного геному, зокрема транспозон IS1 E.coli (інфекція зі стадії 10 т.п.о.) і дублювання 85 п.о., а також елементи, необхідні для розмноження в дріжджах та залишки з сайтів рестрикції[a 1].
Існували суперечки щодо того, чи JCVI-syn1.0 є справжнім синтетичним організмом. Хоча геном синтезувався хімічно у багатьох ділянках, він був сконструйований так, щоб тісно відповідати батьківському геному і пересаджувався в цитоплазму природної клітини. ДНК сама по собі не може створити життєздатну клітину: білки та РНК необхідні для зчитування ДНК, а ліпідні мембрани - для компартменталізації ДНК та цитоплазми. У JCVI-syn1.0 два види, що використовуються як донор і реципієнт, належать до одного роду, зменшуючи потенційні проблеми невідповідності між білками в цитоплазмі хазяїна та новим геномом[a 17]. Пол Кейм (молекулярний генетик з Університету Північної Аризони у Флегстаффі) зазначив, що «попереду великі виклики, перш ніж генні інженери зможуть змішувати, поєднувати та повністю проєктувати геном організму з нуля»[b 4].
Водяні знаки[ред. | ред. код]

Значно розрекламованою особливістю JCVI-syn1.0 є наявність послідовностей «водяних знаків». 4 водяні знаки (показано на Рисунку S1 у додатковому матеріалі статті[a 1]) — це закодовані повідомлення, записані в ДНК, довжиною 1246, 1081, 1109 та 1222 пар основ відповідно. Ці повідомлення не використовували стандартний генетичний код, в якому послідовності 3 основ ДНК кодують амінокислоти, а новий код, винайдений для цієї мети, який читачам було запропоновано розв'язати. Вміст «водяних знаків» такий:
- Водяний знак 1: HTML-скрипт, який читається в браузері як текст, що вітає декодера, та інструкції щодо того, як надіслати авторам електронний лист для підтвердження декодування.
- Водяний знак 2: список авторів та цитата Джеймса Джойса: «Жити, помилятися, падати, тріумфувати, відтворювати життя поза життям».
- Водяний знак 3: більше авторів та цитата Роберта Оппенгеймера (не вказаний): «Бачити речі не такими, як вони є, а такими, якими вони можуть бути».
- Водяний знак 4: більше авторів та цитата Річарда Фейнмана: «Те, що я не можу побудувати, я не можу зрозуміти».
JCVI-syn3.0[ред. | ред. код]
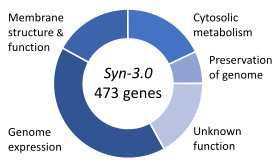
У 2016 році Інститут Вентера використав гени з JCVI-syn1.0 для синтезу меншого геному, який вони називають JCVI-syn3.0, який містить 531560 пар основ і 473 гени[b 7]. У 1996 році, порівнявши M. genitalium з іншою невеликою бактерією Haemophilus influenza, Аркадій Мушегян та Євген Кунін припустили, що може існувати загальний набір з 256 генів, який може бути мінімальним набором генів, необхідним для життєздатності[b 8][a 19]. У цьому новому організмі кількість генів можна зменшити лише до 473, 149 з яких мають функції, які абсолютно невідомі[b 8]. У 2019 році була опублікована повна обчислювальна модель усіх шляхів у клітині Syn3.0, що представляє першу повну модель in silico для живого мінімального організму[a 20].
Проблеми та суперечки[ред. | ред. код]
Сприйняття[ред. | ред. код]
6 жовтня 2007 року Крейг Вентер заявив в інтерв'ю британській газеті The Guardian, що ця ж команда хімічно синтезувала модифіковану версію єдиної хромосоми Mycoplasma genitalium. Синтезований геном ще не був пересаджений в робочу клітину. Наступного дня канадська група з біоетики, ETC Group опублікувала заяву через свого представника, Пата Муні, сказавши, що «творіння» Вентера — це «шасі, на якому можна побудувати майже все. Це може бути внеском у людство, таким як нові ліки або величезна загроза людству, така як біозброя». Вентер прокоментував: «Ми маємо справу з великими ідеями. Ми намагаємось створити нову систему цінностей для життя. Коли маєте справу в такому масштабі, ви не можете очікувати, що всі будуть щасливі»[b 9].
21 травня 2010 року Science повідомив, що група Вентера успішно синтезувала геном бактерії Mycoplasma mycoides з комп'ютерних записів і пересадила синтезований геном в наявну клітину бактерії Mycoplasma capricolum, в якої видалили ДНК. «Синтетична» бактерія була життєздатною, тобто здатною до розмноження[b 1]. Вентер описав його як «перший вид .... що мав батьків комп’ютерів»[b 10].
Створення нової синтетичної бактерії JCVI-3.0 було оголошено в Science 25 березня 2016 року. Вона має тільки 473 гени. Вентер назвав її «першим дизайнерським організмом в історії» й стверджував, що той факт, що 149 необхідних генів мають невідомі функції, означає, що «у всій галузі біології не вистачає третини того, що має важливе значення для життя»[a 21].
Висвітлення в пресі[ред. | ред. код]
Проєкт отримав значне висвітлення у пресі завдяки видовищності Вентера до тієї міри, що Джей Кіслінг, синтетичний біолог-новатор і засновник Amyris сказав, що «Єдиний регламент, який нам потрібен, — це вуста мого колеги»[b 11].
Корисність[ред. | ред. код]
Вентер стверджував, що синтетичні бактерії — це крок до створення організмів для виробництва водню та біопалива, а також для поглинання вуглекислого газу та інших парникових газів. Джордж Макдональд Черч, інший піонер у синтетичній біології, висловив протилежну думку про те, що створення повністю синтетичного геному не є необхідним, оскільки E. coli росте ефективніше, ніж M. genitalium, навіть з усією додатковою ДНК; він прокоментував, що синтетичні гени були включені в E.coli для виконання деяких із зазначених вище завдань[b 12].
Інтелектуальна власність[ред. | ред. код]
Інститут Дж. Крейга Вентера подав заявку на патенти на геном Mycoplasma laboratorium («мінімальний бактеріальний геном») у США та на міжнародному рівні у 2006 році[b 13][b 14][a 22]. Група ETC, канадська група з біоетики, протестувала на тій підставі, що патент був занадто широким за обсягом[b 15].
Подібні проєкти[ред. | ред. код]
З 2002 по 2010 рік команда Угорської академії наук створювала штам кишкової палички під назвою MDS42, який зараз продається Scarab Genomics of Madison, WI під назвою «Clean Genome. E.coli»[b 16], в якому 15% геному батьківського штаму (E. coli K-12 MG1655) було видалено задля підвищення ефективності молекулярно-біологічних досліджень. Видалення IS-елементів, псевдогенів і фагів призводить до кращого збереження кодованих плазмідами токсичних генів, які часто інактивуються транспозонами[a 23][a 24][a 25]. Біохімічні властивості та реплікаційна машинерія не зазнали змін.
Див. також[ред. | ред. код]
Посилання[ред. | ред. код]
Першоджерела[ред. | ред. код]
- ↑ а б в г д Gibson, D. G.; Glass, J. I.; Lartigue, C.; Noskov, V. N.; Chuang, R.-Y.; Algire, M. A.; Benders, G. A.; Montague, M. G.; Ma, L.; Moodie, M. M.; Merryman, C.; Vashee, S.; Krishnakumar, R.; Assad-Garcia, N.; Andrews-Pfannkoch, C.; Denisova, E. A.; Young, L.; Qi, Z.-Q.; Segall-Shapiro, T. H.; Calvey, C. H.; Parmar, P. P.; Hutchison, C. A.; Smith, H. O.; Venter, J. C. (20 травня 2010). Creation of a Bacterial Cell Controlled by a Chemically Synthesized Genome. Science. 329 (5987): 52—56. Bibcode:2010Sci...329...52G. doi:10.1126/science.1190719. PMID 20488990.
- ↑ а б Reich, KA (June 2000). The search for essential genes. Research in Microbiology. 151 (5): 319—24. doi:10.1016/S0923-2508(00)00153-4. PMID 10919511.
- ↑ а б в Glass, John I.; Nacyra Assad-Garcia; Nina Alperovich; Shibu Yooseph; Matthew R. Lewis; Mahir Maruf; Clyde A. Hutchison; Hamilton O. Smith; J. Craig Venter (10 січня 2006). Essential genes of a minimal bacterium. Proceedings of the National Academy of Sciences. 103 (2): 425—430. Bibcode:2006PNAS..103..425G. doi:10.1073/pnas.0510013103. PMC 1324956. PMID 16407165.
- ↑ Gibson, D. G.; Benders, G. A.; Andrews-Pfannkoch, C.; Denisova, E. A.; Baden-Tillson, H.; Zaveri, J.; Stockwell, T. B.; Brownley, A.; Thomas, D. W. (29 лютого 2008). Complete Chemical Synthesis, Assembly, and Cloning of a Mycoplasma genitalium Genome. Science (англ.). 319 (5867): 1215—1220. Bibcode:2008Sci...319.1215G. doi:10.1126/science.1151721. ISSN 0036-8075. PMID 18218864.
- ↑ Hutchison CA, Montague MG (2002). Mycoplasmas and the minimal genome concept. Molecular Biology and Pathogenicity of Mycoplasmas (Razin S, Herrmann R, eds.). New York: Kluwer Academic/Plenum. с. 221–54. ISBN 978-0-306-47287-9.
- ↑ Young L, Sung J, Stacey G, Masters JR. "Detection of Mycoplasma in cell cultures". Nat Protoc. 2010 5(5): 929–34. Epub 2010 Apr 22.
- ↑ Fraser CM, Gocayne JD, White O та ін. (October 1995). The minimal gene complement of Mycoplasma genitalium. Science. 270 (5235): 397—403. Bibcode:1995Sci...270..397F. doi:10.1126/science.270.5235.397. PMID 7569993.
- ↑ Morris RM та ін. (2002). SAR11 clade dominates ocean surface bacterioplankton communities. Nature. 420 (6917): 806—10. Bibcode:2002Natur.420..806M. doi:10.1038/nature01240. PMID 12490947.
- ↑ Stephen J. Giovannoni; H. James Tripp та ін. (2005). Genome Streamlining in a Cosmopolitan Oceanic Bacterium. Science. 309 (5738): 1242—1245. Bibcode:2005Sci...309.1242G. doi:10.1126/science.1114057. PMID 16109880.
- ↑ Rappé MS, Connon SA, Vergin KL, Giovannoni SL (2002). Cultivation of the ubiquitous SAR11 marine bacterioplankton clade. Nature. 418 (6898): 630—33. Bibcode:2002Natur.418..630R. doi:10.1038/nature00917. PMID 12167859.
- ↑ Tripp HJ, Kitner JB, Schwalbach MS, Dacey JW, Wilhelm LJ, Giovannoni SJ (10 квітня 2008). SAR11 marine bacteria require exogenous reduced sulphur for growth. Nature. 452 (7188): 741—4. Bibcode:2008Natur.452..741T. doi:10.1038/nature06776. PMID 18337719.
- ↑ Nakabachi, A.; Yamashita, A.; Toh, H.; Ishikawa, H.; Dunbar, H. E.; Moran, N. A.; Hattori, M. (2006). The 160-Kilobase Genome of the Bacterial Endosymbiont Carsonella. Science. 314 (5797): 267. doi:10.1126/science.1134196. PMID 17038615.
- ↑ McCutcheon, J. P.; McDonald, B. R.; Moran, N. A. (2009). Convergent evolution of metabolic roles in bacterial co-symbionts of insects. Proceedings of the National Academy of Sciences. 106 (36): 15394—15399. Bibcode:2009PNAS..10615394M. doi:10.1073/pnas.0906424106. PMC 2741262. PMID 19706397.
- ↑ Nancy A. Moran and Gordon M. Bennett (2014). The Tiniest Tiny Genomes. Annual Review of Microbiology. 68: 195—215. doi:10.1146/annurev-micro-091213-112901. PMID 24995872.
- ↑ Lartigue C, Glass JI, Alperovich N, Pieper R, Parmar PP, Hutchison CA 3rd, Smith HO, Venter JC (Aug 3, 2007). Genome transplantation in bacteria: changing one species to another. Science. 317 (5838): 632—8. Bibcode:2007Sci...317..632L. doi:10.1126/science.1144622. PMID 17600181.
- ↑ а б Gibson, B; Clyde A. Hutchison; Cynthia Pfannkoch; J. Craig Venter та ін. (24 січня 2008). Complete Chemical Synthesis, Assembly, and Cloning of a Mycoplasma genitalium Genome. Science. 319 (5867): 1215—20. Bibcode:2008Sci...319.1215G. doi:10.1126/science.1151721. PMID 18218864.
- ↑ Povolotskaya, IS; Kondrashov, FA (Jun 2010). Sequence space and the ongoing expansion of the protein universe. Nature. 465 (7300): 922—6. Bibcode:2010Natur.465..922P. doi:10.1038/nature09105. PMID 20485343.
- ↑ Hutchison, Clyde A.; Chuang, Ray-Yuan; Noskov, Vladimir N.; Assad-Garcia, Nacyra; Deerinck, Thomas J.; Ellisman, Mark H.; Gill, John; Kannan, Krishna; Karas, Bogumil J. (25 березня 2016). Design and synthesis of a minimal bacterial genome. Science (англ.). 351 (6280): aad6253. Bibcode:2016Sci...351.....H. doi:10.1126/science.aad6253. ISSN 0036-8075. PMID 27013737.
- ↑ Arcady R. Mushegian and Eugene V. Koonin (September 1996). A minimal gene set for cellular life derived by comparison of complete bacterial genomes. Proc. Natl. Acad. Sci. USA. 93 (19): 10268—10273. Bibcode:1996PNAS...9310268M. doi:10.1073/pnas.93.19.10268. PMC 38373. PMID 8816789.
- ↑ Breuer, Marian; Earnest, Tyler M.; Merryman, Chuck; Wise, Kim S.; Sun, Lijie; Lynott, Michaela R.; Hutchison, Clyde A.; Smith, Hamilton O.; Lapek, John D.; Gonzalez, David J.; De Crécy-Lagard, Valérie; Haas, Drago; Hanson, Andrew D.; Labhsetwar, Piyush; Glass, John I.; Luthey-Schulten, Zaida (2019). Essential metabolism for a minimal cell. eLife. 8. doi:10.7554/eLife.36842. PMC 6609329. PMID 30657448.
{{cite journal}}: Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання) - ↑ Herper, Matthew. After 20 Year Quest, Biologists Create Synthetic Bacteria With No Extra Genes. Forbes (англ.). Архів оригіналу за 24 січня 2021. Процитовано 2 липня 2019.
- ↑ US Patent Application: 20070122826 [Архівовано 25 листопада 2021 у Wayback Machine.]
- ↑ Umenhoffer K, Fehér T, Balikó G, Ayaydin F, Pósfai J, Blattner FR, Pósfai G (2010). Reduced evolvability of Escherichia coli MDS42, an IS-less cellular chassis for molecular and synthetic biology applications. Microbial Cell Factories. 9: 38. doi:10.1186/1475-2859-9-38. PMC 2891674. PMID 20492662.
{{cite journal}}: Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання) - ↑ Pósfai G, Plunkett G 3rd, Fehér T, Frisch D, Keil GM, Umenhoffer K, Kolisnychenko V, Stahl B, Sharma SS, de Arruda M, Burland V, Harcum SW, Blattner FR (2006). Emergent properties of reduced-genome Escherichia coli. Science. 312 (5776): 1044—6. Bibcode:2006Sci...312.1044P. doi:10.1126/science.1126439. PMID 16645050.
- ↑ Kolisnychenko V, Plunkett G 3rd, Herring CD, Fehér T, Pósfai J, Blattner FR, Pósfai G (April 2002). Engineering a reduced Escherichia coli genome. Genome Res. 12 (4): 640—7. doi:10.1101/gr.217202. PMC 187512. PMID 11932248.
Популярна преса[ред. | ред. код]
- ↑ а б Roberta Kwok (2010). Genomics: DNA's master craftsmen. Nature. 468 (7320): 22—5. Bibcode:2010Natur.468...22K. doi:10.1038/468022a. PMID 21048740.
- ↑ а б в Callaway, Ewen (2016). 'Minimal' cell raises stakes in race to harness synthetic life. Nature (англ.). 531 (7596): 557—558. Bibcode:2016Natur.531..557C. doi:10.1038/531557a. ISSN 0028-0836. PMID 27029256.
- ↑ Ball, Philip (24 січня 2008). Genome stitched together by hand. Nature (англ.). doi:10.1038/news.2008.522. ISSN 0028-0836. Архів оригіналу за 23 січня 2022. Процитовано 3 лютого 2021.
- ↑ а б в г Pennisi E (May 2010). Genomics. Synthetic genome brings new life to bacterium (PDF). Science. 328 (5981): 958—9. doi:10.1126/science.328.5981.958. PMID 20488994. Архів оригіналу (PDF) за 25 травня 2010. Процитовано 3 лютого 2021.
- ↑ Katsnelson, Alla (20 травня 2010). Researchers start up cell with synthetic genome. Nature (англ.). doi:10.1038/news.2010.253. ISSN 0028-0836. Архів оригіналу за 23 січня 2022. Процитовано 3 лютого 2021.
- ↑ Zimmer, Carl (23 серпня 2013). And the Genomes Keep Shrinking... National Geographic. Архів оригіналу за 20 вересня 2017. Процитовано 3 лютого 2021.
- ↑ First Minimal Synthetic Bacterial Cell. Astrobiology Web. March 24, 2016.
- ↑ а б Yong, Ed (24 березня 2016). The Mysterious Thing About a Marvelous New Synthetic Cell. Архів оригіналу за 1 лютого 2021. Процитовано 3 лютого 2021.
- ↑ Pilkington, Ed (6 жовтня 2009). I am creating artificial life, declares US gene pioneer. London: The Guardian. Архів оригіналу за 28 травня 2010. Процитовано 23 листопада 2012.
- ↑ How scientists made 'artificial life'. BBC News. 20 травня 2010. Архів оригіналу за 1 червня 2013. Процитовано 21 травня 2010.
- ↑ Andrew Pollack, His Corporate Strategy: The Scientific Method, NYTimes, September 4, 2010
- ↑ Longest Piece of Synthetic DNA Yet [Архівовано 16 листопада 2008 у Wayback Machine.], Scientific American News, 24 January 2008
- ↑ "Artificial life: Patent pending [Архівовано 3 жовтня 2008 у Wayback Machine.]", The Economist, June 14, 2007. Retrieved October 7, 2007.
- ↑ Roger Highfield, "Man-made microbe 'to create endless biofuel' [Архівовано 10 квітня 2008 у Wayback Machine.]", Telegraph, June 8, 2007. Retrieved October 7, 2007.
- ↑ Ianculovici, Elena (15 січня 2008). The Emerging Field of Synthetic Biology: "Syn" or Salvation?. Science in the News (амер.). Архів оригіналу за 14 лютого 2021. Процитовано 3 липня 2019.
- ↑ Scarab Genomics LLC. Company web site. Архів оригіналу за 8 липня 2021. Процитовано 3 лютого 2021.
Джерела[ред. | ред. код]
|