Процесинг РНК

Проце́синг, котранскрипці́йна модифіка́ція, посттранскрипці́йна модифіка́ція — дозрівання новосинтезованої молекули РНК до її функціонально активної форми.
Яскравим прикладом процесингу є дозрівання пре-мРНК до зрілої мРНК, із якої в цитоплазмі буде зчитуватися інформація про амінокислотну послідовність білків (трансляція). Утім, процесингу зазнають не лише мРНК, а й багато видів некодуючих РНК, транспортна РНК та рибосомна РНК.
У підручниках часто пишуть, що дозрівання пре-мРНК відбувається після її зчитування з ДНК матриці[2]. Таке явище відповідає лабораторним умовам (in vitro), коли стадії процесингу мРНК вивчають поступово, незалежно одна від одної. Але за умов in vivo, у живих клітинах, процесинг мРНК відбувається безпосередньо під час транскрипції в складі РНК-полімеразного комплексу[1]. Тому коректним терміном для мРНК дозрівання є котранскрипційна модифікація[2].
Проте не всі види РНК зазнають процесингу під час синтезу. Так, молекули тРНК дріжджів Saccharomyces cerevisiae дозрівають (зазнають сплайсингу) у цитоплазмі з подальшим експортом у ядро і поверненням до цитоплазми[3], тому в такому випадку коректним є термін «посттранскрипційна модифікація». Але у фаховій літературі частіше використовують термін «процесинг тРНК».
Процесинг мРНК[ред. | ред. код]

Процесинг пре-мРНК до зрілої мРНК відбувається безпосередньо в складі РНК-полімеразного комплексу в ядрі. Синтез мРНК виконує РНК-полімераза II. До її C-кінцевого домену приєднуються фактори, що діють на різних стадіях дозрівання транскрипту. Оскільки процесинг відбувається безпосередньо під час синтезу молекули РНК, а сплайсинг відбувається одразу після синтезу сплайс-сайтів, то пре-мРНК транскрипт для більшості генів багатоклітинних організмів можна назвати умовною теоретичною молекулою, яка не існує як така in vivo[4]. Однак можливі варіанти сплайсингу поза РНК-полімеразним комплексом[5].
Загалом процесинг мРНК ділиться на зміну 3'- та 5'-кінцевих ділянок молекули (кепування та поліаденілування, відповідно) та сплайсинг (вирізання частин молекули РНК — інтронів). Модифікації 3'- та 5'-кінцевих ділянок молекули РНК, не лише мРНК, дуже важливі для стабільності РНК під час перебування в ядрі. «Відкриті» кінці РНК стають мішенями нуклеаз, таких як комплекс екзосома. Тому молекули РНК, які перебувають певний час у ядрі мають зміни на своїх 3'- та 5'-кінцевих ділянках.[6]
Кепування[ред. | ред. код]

Кепування — це додавання кепу до 5'-кінця[en] пре-мРНК, що відбувається в декілька стадій за допомогою ферменту, що приєднує кеп (англ. mRNA-capping enzyme), та гуаніл-N7-метилтрансферази. Унаслідок цих реакцій на 5'-кінці мРНК формуються один чи два модифіковані нуклеотиди (найчастіше — метил-гуанідин), з'єднані з рештою мРНК незвичайним 5'-5' трифосфатним зв'язком.
Кепування виконує такі функції:
- захист мРНК від деградації нуклеазами;
- участь у подальшому процесингу, сплайсингу;
- експорт мРНК у цитоплазму;
- участь у трансляції.
Сплайсинг[ред. | ред. код]
Сплайсинг — це механізм, завдяки якому вирізаються інтрони — некодуючі ділянки пре-мРНК, тобто послідовності, які потім не будуть використані як матриця для біосинтезу білків. Зріла мРНК втрачає інтрони, залишаючи у своєму складі лише екзони. Сплайсинг відбувається за участі сплайсосоми, що містить малі ядерні РНК. У багатьох випадках пре-мРНК ще продовжує синтезуватися, тоді як інтрони вже вирізаються. Інтрон, що вирізається, має форму ласо (англ. lariat). Швидкість сплайсингу в евкаріот досить велика: у 10 % випадків у дріжджів сплайсосома може відрізати інтрон, коли полімераза знаходиться на відстані лише у 26-36 нуклеотидів, а 50 % сплайсингу завершується за 1,4 сек. після завершення синтезу 3'-кінцевого сплайс-сайту[7].
Альтернативний сплайсинг[ред. | ред. код]

У багатьох еукаріотів кількість білків, що можуть формуватись у клітинах, значно перевищує кількість генів, закодованих у ядрі. Так, згідно з результатами проєкту ENCODE (Енциклопедія елементів ДНК) 2012 року[9], у людини наявний 21 061 ген, що кодує білки, тоді як самих білків у клітинах людини — від 250 000 до мільйона. Різноманіття білків порівняно з кількістю генів досягається також завдяки явищу альтернативного сплайсингу.
При альтернативному сплайсингу з молекули пре-мРНК вирізаються різні комбінації інтронів, а перекомбіновані екзони зшиваються та формують різні зрілі мРНК. Таким чином, з одного гена можна отримати одну пре-мРНК, але багато видів зрілої мРНК і, відповідно, багато різних білків.
Гени пивних дріжджів Saccharomyces cerevisiae містять невелику кількість коротких інтронів, а в інших одноклітинних еукаріот, наприклад, трипаносом, найчастіше їх зовсім немає. Тому альтернативний сплайсинг в одноклітинних ядерних відбувається рідко[4].
Натомість у багатоклітинних еукаріотів альтернативний сплайсинг — дуже поширене явище. Так, у людини приблизно 95 % пре-мРНК підлягають цьому процесу[10]. Варіанти мРНК, що утворилися з однієї пре-мРНК, називають сплайс-варіантами або мРНК-ізоформами[11], і часто вони бувають тканинно-специфічними (тобто один сплайс-варіант існує в одній тканині, другий — в іншій). У плодової мухи D. melanogaster альтернативний сплайсинг спричинює детермінацію статі, а один ген Dscam (англ. Down syndrome cell adhesion molecule) кодує понад 38 000 різних варіантів мРНК і, відповідно, білків (кількість, що більша за число генів у цього виду мух)[4].
Основними регуляторам вибору варіантів сплайсингу є сила сплайс-сайту, модифікації гістонових хвостів (див. ілюстрацію «Схематичне зображення котранскрипційного процесингу пре-мРНК до зрілої мРНК») та позиція нуклеосоми[4], взаємодія РНК з різними РНК-зв'язуючими білками тощо[12]. Сила сплайс-сайту відповідає частоті впізнавання цього сайту сплайсосомою і включення даного екзону у всіх синтезованих мРНК — це так званий «конститутивний сплайсинг»[12]. У регуляції сплайсингу беруть участь білки регулятори сплайсингу (англ. SR proteins), такі як NOVA1[13] та PTBP1 (англ. Polypyrimidine tract-binding protein 1)[14] та гетерогенні ядерні рибонуклеопротеїни (гяРНП, англ. hnRNP)[11]
Окрім участі альтернативного сплайсингу в нормальній життєдіяльності організму, це явище може призводити до хвороб.[15]
Проте не всі транскрипти, згенеровані альтернативним сплайсингом, можуть кодувати білки чи виконувати інші функції.[12]
Поліаденілування[ред. | ред. код]
Після відрізання (англ. cleavage) транскрипту від РНК-полімеразного комплексу до молекули пре-мРНК із 3'-кінця[en] додається хвіст із багатьох залишків аденіну, звідки походить і назва реакції. Сигнал поліаденілування (поліА- чи polyA-сигнал, іноді polyA-сайт), так само як і стартовий сигнал, закодований у гені, і, відповідно, зчитується РНК полімеразою II у пре-мРНК. PolyA-сигнал має 2 компоненти: послідовність із 6 нуклеотидів AAУАAA (Т у ДНК замінюється на У в РНК), та У- або Г/У-збагачена послідовність, що розміщується на відстані 20 нуклеотидів від першого сигналу. Білки CPSF (англ. Cleavage-Polyadenylation Specificity Factor) та CstF (англ. Cleavage stimulation Factor) розпізнають ці сигнали[16].
PolyA-полімераза синтезує хвіст довжиною в 100—200 аденінових нуклеотидів. Довжина polyA-хвоста варіює між різними видами. Так, у людини в середньому додається 250—300 аденінів, а у дріжджів — 70-80[16].
Поліаденілування відбувається одразу після розщеплення молекули РНК (червоні ножиці на схемі «Котранскрипційний процесинг пре-мРНК до зрілої мРНК»), тому сигнал для поліаденілування ще називають сигналом для розщеплення та поліаденілування (англ. cleavage and polyadenylation)[17].
Поліаденілування виконує такі функції[17]:
- Експортування мРНК з ядра;
- Стабільність мРНК, включаючи захист мРНК від деградації нуклеазами;
- Участь у трансляції;
- Посттранскрипційна регуляція[en] експресії генів. Довжина polyA хвоста має важливе значення для контролю кількості білків, що будуть синтезовані із цієї РНК. Також у 3'-нетрансльованій ділянці містяться сайти розпізнавання молекулами РНК інтерференційного механізму, такими як мікроРНК.
Альтернативне поліаденілування[ред. | ред. код]
Наприкінці першого десятиріччя XXI ст. стало можливим робити секвенування РНК[en] (англ. RNA-Seq) цілих транскриптомів. Це дало можливість встановити, що багато генів людини мають більше одного polyA-сигналу, що призводить до формування різних 3'-кінців однієї пре-мРНК[16].
До 2011 року стало зрозуміло, що багато білок-кодуючих генів мають не один, а декілька сайтів поліаденілування, між якими відбувається вибір під час синтезу мРНК. У людини 50 % генів можуть кодувати різні транскрипти завдяки альтернативному поліаденілуванню[18].
У деяких випадках альтернативне поліаденілування призводить до зміни амінокислотної послідовності білків, тоді як в інших варіації виникають в 3'-некодуючій ділянці мРНК, а кодуюча послідовність залишається незмінною[18]. При виборі сайту поліаденілування може трапитись вкорочення 3'-нетрансльованої ділянки молекули мРНК, що призведе до втрати регуляторних елементів для взаємодії з мікроРНК[19], тоді, найімовірніше, мРНК буде менше пригнічуватися, і відповідно, з неї синтезуватиметься більше білків[18].
Вибір polyA-сайту залежить від багатьох умов, таких як ріст клітини, її розвиток та диференціація, а також патологічні процеси, зокрема розвиток пухлин[18].
Прикладом альтернативного поліаденілування є ген важкого ланцюга імуноглобуліну IgM, білок якого проходить зміну від мембраннозв'язаної до вільної форми. Така зміна є результатом вибору одного із двох polyA-сайтів[18].
Альтернативне поліаденілування часто трапляється під час утворення нейронів, де за рахунок такого процесу сотні генів мають більші 3' нетрансльовані ділянки — такі гени отримали назву подовжених (англ. extended gene). Основним білком, який призводить до формування довшого 3'-кінця у нейронів — РНК-зв'язуючий білок Elav (англ. embryonic lethal abnormal visual). У плодової мухи його активність залежить від промоторної ділянки генів. У подовжених генів РНК-полімераза II — фермент, що синтезує РНК — затримується на довший час на початкових стадіях транскрипції в порівнянні з іншими генами[20][21].
Цікавим є те, що альтернативне поліаденілування може впливати на розміщення білку, що зчитується з даної мРНК у клітині, хоча при цьому не змінюється просторове розміщення самої мРНК у цитоплазмі. Так мРНК CD47 у клітинних лініях людини може мати два варіанти 3'-нетрансльованої ділянки: коротшу і довшу. Білки CD47, які транслювались із мРНК з довшою 3'-нетрансльованою ділянкою, розміщувалися здебільшого біля поверхні клітин, а CD47-протеїни, які було синтезовано з коротшої форми мРНК, були знайдені в ендоплазматичному ретикулумі[22].
- Відсутність поліаденілування, полі-A(-)
мРНК гістонів типово не підлягають поліаденілуванню у всіх організмів, натомість мають специфічні структури на 3'-кінці (див. п «Процесинг мРНК гістонів»), які захищають їх від деградації нуклеазами. Але крім мРНК гістонів у клітинних лініях H9 та HeLa було знайдено мРНК, які можуть у деяких випадках не мати полі-A хвосту, такі як мРНК znf460 та sesn3[23]
Регуляція процесингу мРНК[ред. | ред. код]
Паузи, які робить РНК-полімераза II, модифікації її C-кінцевого домену (фосфорилювання), метилювання ДНК, позиція нуклеосоми, модифікації гістонових хвостів (їх метилювання[en], ацетилювання та деацетилювання[en]), формування вторинних структур синтезованої РНК[24], модифікації нуклеотидів та редагування РНК — всі ці фактори, що, по-перше, можуть впливати один на одного, а по-друге регулюють процесинг, призводять до наявності чи відсутності кепу, вибору сайтів сплайсингу та polyA-сайтів для альтернативного сплайсингу та поліаденілування, відповідно. Таким чином ці зміни призводять до різноманіття мРНК та встановлюють долю транскрипту: чи буде мРНК одразу руйнуватися екзосомним комплексом у ядрі, чи буде експортована до цитоплазми і чи зможуть мікроРНК взаємодіяти з нею, пригнічуючи біосинтез білків та/або деградувати її в цитоплазмі[2].
Процесинг мРНК гістонів[ред. | ред. код]

Синтез і дозрівання мРНК гістонів, що складають нуклеосому (H2A, H2B, H3 та H4) і лінкерного гістону H1 відрізняється від процесингу інших еукаріотичних мРНК. По-перше синтез мРНК цих гістонів відбувається у строгій залежності від клітинного циклу — гени гістнонів експресуються на початку S-фази. При чому по завершенню S-фази або при припиненні з різних причин реплікації ДНК, мРНК цих гістонів швидко руйнується комплексом екзосоми[25].
Структура мРНК зазначених п'яти гістонів відрізняється від мРНК інших генів — це єдині відомі мРНК еукаріот, у яких стабільно відсутній polyA-хвіст, тобто у них не відбувається поліаденілування. Натомість мРНК гістонів мають структуру стебло-петля, яка формується 6 парами нуклеотидів та 4 нуклеотидами у самій петлі. Також особливістю структури мРНК гістонів є відсутність інтронів та невеликі розміри 5'- та 3'-нетрансльованих ділянок, хоча це може бути притаманним і іншим еукаріотичним генам[25].
Оскільки структура мРНК гістонів відрізняється від мРНК інших генів, процесинг їх також має особливості. Сплайсинг мРНК гістонів не відбувається за відсутності інтронів. Процесинг 3'-нетрансльованої ділянки включає в себе низку нестандартних ферментів та рибонуклеопротеїнових комплексів, таких як U7 мяРНК, ZFP100[26], LSM11-LSM10[27][28]. Відрізання мРНК гістону під час транскрипції відбувається у місці між структурою стебло-петля і послідовністю, яка знаходиться на відстані 15 нуклеотидів далі і має назву HDE (англ. histone downstream element)[25].
Цікавим є те, що варіанти гістонів, такі як H3.3, macro-H2A, H2A.Z та H1.0 синтезуються та процесуються як інші мРНК. Тобто їх транскрипція йде не лише в S-фазу, а впродовж всього циклу клітини, та в них відбувається поліаденілування[25].
Процесинг тРНК[ред. | ред. код]

Процес біосинтезу білків має необхідну ланку — молекулу транспортної РНК (тРНК), яка повинна розпізнати триплет — послідовність із трьох нуклеотидів матричної РНК (мРНК), що завантажена на рибосому, і відповідно до такого триплетного коду донести амінокислоту до поліпептидного ланцюга білку, що синтезується.
Але молекула тРНК не синтезується в готовому вигляді — вона транскрибується РНК-полімеразою III[en] з ДНК матриці у вигляді пре-тРНК і проходить стадію дозрівання, у результаті якої набуває третинної структури L-подібної форми, завдовжки у 74-95 нуклеотидів (найчастіше 76). З одного кінця вона містить антикодонову послідовність, що буде комплементарною кодону мРНК, з іншого — амінокислоту (акцепторне стебло)[29].
Процесинг тРНК включає п'ять етапів[30]:
- Від'єднання 5'-послідовності (англ. 5′ leader) РНКазою P[en] за участі рибонуклеопротеїнового комплексу.
- Від'єднання 3'-хвоста (англ. 3′ trailer) комбінацією з декількох екзо- та ендонуклеаз.
- Додавання послідовності трьох нуклеотидів CCA (цитозин-цитозин-аденін) до 3' кінця молекули, які будуть неспареними і до яких буде приєднуватися амінокислота.
- Вирізання інтрону (сплайсинг), яке відбувається в більшості еукаріотів та у деяких тРНК архей.
- Модифікації тРНК в різних місцях включаючи редагування РНК, метилювання нуклеотидів.
5'-кінець пре-тРНК розрізається РНКазою P, а 3'-послідовність процесується тРНКазою Z (англ. tRNase Z) та іншими ферментами[31].
У людини трапляються пре-тРНК як з інтроном, так і без нього. Ендонуклеаза, яка здійснює сплайсинг пре-тРНК, має чотири компоненти, що формують комплекс TSEN (англ. tRNA splicing endonuclease, TSEN, SEN)[32]: каталітичні субодиниці TSEN2 та TSEN34 й структурні субодиниці TSEN15 та TSEN54[33]. Ці субодиниці виникли внаслідок кількох випадків дуплікації генів у ході еволюції ядерних, із подальшою спеціалізацією кожної субодиниці. Sen2 та Sen34 субодиниці мають найбільший рівень гомології з нуклеазами архей[34].
Процесинг тРНК сильно відрізняється у різних організмів — для кожного із п'яти етапів у ході еволюції виникли різні сценарії: декілька можливостей формування 3'- та 5'-кінців, два різних шляхи сплайсингу тРНК, варіації у додаванні CCA і механізмах контролю якості тРНК. Додатковими варіаціями є декілька шляхів експорту тРНК із цитоплазми в ядро, також був відкритий механізм імпорту тРНК в мітохондрії[30]. У мітохондріях всіх організмів відбувається власна трансляція, але мітохондріальна ДНК містить не всі гени тРНК, отже деякі з них повинні доставлятись спочатку з ядра в цитоплазму, а далі із цитоплазми в мітохондрію.
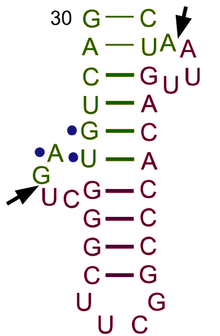
У більшості організмів основним етапом вирізання інтрону тРНК є розпізнавання специфічної структури BHB (англ. bulge-helix-bulge), яка є маркером інтрон-екзонного переходу і складається з таких частин: коротка послідовність неспарених нуклеотидів, потім спіраль, сформована спареними нуклеотидами, за нею — знову коротка послідовність неспарених нуклеотидів (див. ілюстрацію).
У червоних водоростей Cyanidioschyzon merolae[en] є гени тРНК, що кодують транскрипт із декількома інтронами, або такими інтронами, де 3'-частина кодуючої послідовності тРНК лежить в 5'-частині гена. Для процесингу таких пре-тРНК кінці молекули повинні бути зшиті у кільце, і вже потім відбуається сплайсинг[36].
Процесинг рРНК[ред. | ред. код]
Молекули рРНК формують коровий комплекс (серцевину) рибосоми. Утворення більшості варіантів пре-рРНК відбувається в ядерцях, де міститься кілька тандемних повторів генів рРНК. РНК-полімераза I[en] синтезує з матриці ДНК довгий продукт, поліцистронну РНК (англ. polycistronic RNA), пре-рРНК. Потім вона розрізається на окремі молекули рРНК. На відміну від інших типів рРНК, 5S рРНК синтезуються в ядрі поза ядерцем і цей процес каталізує РНК-полімераза III[en][37][38].
Процесинг рРНК дуже консервативний у більшості організмів і складається з таких стадій[39]:
- транскрипції пре-рРНК у вигляді довгої поліцистронної РНК, та додаткових окремих рРНК;
- модифікації ділянок пре-рРНК;
- розрізання пре-рРНК до зрілих рРНК;
- формування комплексу з рибосомними білками;
- в еукаріотів додаткові стадії включають у себе імпорт рибосомних білків із цитоплазми в ядро та подальший експорт рибосомних субодиниць у цитоплазму.
Модифікація та сплайсинг пре-рРНК відбувається завдяки одному з типів некодуючих РНК — малим ядерцевим РНК[en], мяцРНК (англ. snoRNA), що взаємодіють із малими ядерцевими білками (англ. snoRNP) в еукаріотів та малими рибонуклеопротеїнами (англ. sRNP) в архей. Потім 5,8[en] та 28S рРНК[en] взаємодіють із комплексом 5S рРНК та L5 рибосомним білком та іншими рибосомними білками і формують 60S субодиницю (у еукаріот), а 18S рРНК формує 40S субодиницю рибосоми. 40S та 60S рибосомні субодиниці експортуються до цитоплазми, де вони з'єднуються з мРНК і формують рибосому[37].
Процесинг 5S рРНК у дріжджів відбувається за допомогою екзонуклеаз Rex1p, Rex2p, й Rex3p та Ro білка. 5S рРНК може бути поліаденільованна та деградована комплексом екзосоми[40].
В архей, які найчастіше мають одну копію кожного гена, можуть бути більше ніж вісім копій генів рРНК. Гени рРНК транскрибуються поліцистронно в одному опероні, але у деяких організмів, таких як H. cutirubrum гени рРНК перемішані з генами тРНК в одному опероні. Тоді як у Thermoproteus tenax[en] та Desulfurococcus mobilis[en] 5S рРНК ген не закодований в одному опероні з іншими рРНК — він повинен бути транскрибований незалежно[41].
Видалення інтронів в архей відбувається завдяки нуклеазам, які специфічні до цього домену організмів і основним моментом є розпізнавання мотиву, що є маркером екзон-інтронного переходу BHB (англ. bulge-helix-bulge) як і у сплайсингу тРНК (див ілюстрацію «BHB мотив сплайсингу тРНК»)[41].
Процесинг інших некодуючих РНК[ред. | ред. код]
Некодуючі РНК, нкРНК — це функціональні РНК молекули, нуклеотидна послідовність яких не переводиться в амінокислотну послідовність білків, звідси і назва — вони не кодують білки. Функції нкРНК полягають в регуляції експресії генів на різних рівнях (транскрипція, сплайсинг, мРНК деградація, трансляція), вплив на структуру хроматину. Некодуючі РНК бувають короткими або малими (англ. small ncRNA), такими як мікроРНК чи піРНК, та довгими нкРНК, що більше за 200 нуклеотидів у довжину, наприклад Xist, що бере участь в інактивації X-хромосоми[42].
Некодуючі РНК, що беруть участь у РНК інтерференції[ред. | ред. код]

РНК-інтерференція, РНКі, (RNA interference, RNAi) — механізм, що регулює експресію генів в еукаріотів шляхом деградації цілевої мРНК та/або приглушення трансляції. У РНКі беруть участь дволанцюгові малі некодуючі РНК, що можуть походити з генів, некодуючих частин ДНК, антисенс РНК чи інвертованих повторів[en]. Також малі нкРНК можуть потрапляти в клітину екзогенно, з вірусів, призводячи до клітинної загибелі шляхом апоптозу — способу позбавитися заражених клітин в організмі[47]. Такі короткі некодуючі РНК синтезуються у вигляді прекурсорів (попередників), які повинні пройти стадію процесингу для того, щоб сформувати зрілі, функціональні РНК, які будуть взаємодіяти з РНК-індукованим комплексом заглушення (RISC англ. , RNA induced silencing complex) і впливати на експресію цільових генів.
Процесинг мікроРНК[ред. | ред. код]
МікроРНК походять із частин ДНК, які можуть кодувати власне лише мікроРНК, із мікроРНК кластерів, можуть міститися в інтронах генів, що кодують білки, та бути мітронами — частинами інтронів мРНК, що процесуються за допомогою сплайсосоми та комплексу екзосома, а не ферменту Дроша[48]. Одразу після транскрипції мікроРНК прекурсори називаються прі-мікроРНК і мають свою власну, характерну структуру, стебло-петля (англ. stem–loop) зі шпилькою, оточеною послідовністю дволанцюгової РНК. Прі-мікроРНК розпізнається ферментом Дроша, який відрізає нуклеотиди по боках від структури стебло-петля, формуючи пре-мікроРНК, довжиною приблизно 70 нуклеотидів[48]. Пре-мікроРНК переноситься з ядра до цитоплазми за допомогою експортину-5[49]. У цитоплазмі фермент Дайсер відрізає шпилькову частину пре-мікроРНК, формуючи дволанцюгову, не повністю комплементарно-зв'язану структуру довжиною 21-24 нуклеотиди — мікроРНК/мікроРНК* (з зірочкою). Далі білок родини Аргонавт обирає мікроРНК з дуплекса, а мікроРНК* деградується. Зріла мікроРНК взаємодіє з RISC і призводить до деградації/заглушення трансляції із цільової мРНК, до якої дана мікроРНК частково комплементарна. Сайти взаємодії з мікроРНК частіше лежать у 3' нетрансльованій ділянці мРНК (англ. 3' UTR), але також і в їх екзонах[50].
Процесинг міРНК[ред. | ред. код]
Малі інтерферуючі РНК вперше були вивчені на рослинах, як похідні вірусів. Зараз зрозуміло, що міРНК в еукаріотів походять із різних частин геному, і можуть бути екзогенного походження. Біогенез міРНК залежить від того, чи для цього необхідна РНК-залежна РНК-полімераза[51].
У тварин міРНК формуються з дволанцюгових РНК (длРНК, англ. dsRNA). У цитоплазмі (або ядрі) Дайсер виконує розрізання длРНК на міРНК-дуплекс довжиною 20-25 нуклеотидів із 2 нуклеотидами, неспареними на 3' кінці, та 5' монофосфатом. Один із двох ланцюгів міРНК-дуплексу взаємодіє з білком сімейства Аргонавт, і цей комплекс призводить до РНК-індукованого заглушення генів за участі RISC[46].
У рослин та червів формування міРНК залежить від РНК-залежної РНК-полімерази (RdRP). У рослин формується прекурсор міРНК, другий, комплементарний ланцюг якого синтезується за допомогою RdRP. У результаті дволанцюгова РНК розрізається за допомогою Дайсера на міРНК, які метилюються ферментом HEN1 і взаємодіють із білками родини Аргонавт[51].
У Caenorhabditis elegans прекурсори міРНК формуються з довгих дволанцюгових РНК за допомогою ферменту Дайсер (DCR-1), і взаємодіють із білком родини Аргонавт. Такий комплекс з'єднується із цільовою мРНК і за допомогою РНК-залежної РНК-полімерази синтезуються вторинні міРНК з 5' трифосфатними кінцями[51].
Різниця між функціонуванням малих інтерферуючих РНК та мікроРНК — це повна чи неповна комплементарність даної нкРНК до послідовності мРНК, відповідно. При взаємодії мікроРНК з мРНК деякі нуклеотиди залишаються неспареними. Вважається, що міРНК в природі частіше зустрічаються у рослин[52].
Процесинг піРНК[ред. | ред. код]
Більшість білків сімейства Аргонавт взаємодіють як із міРНК так і з мікроРНК, але є підродина, PIWI (англ. P-element-induced wimpy testis), що специфічно функціонує з піРНК для заглушення активності транспозонів[52].
Зчитуються піРНК з піРНК-кластерів або активних транспозонів. Обидва види транскриптів досить довгі і для дозрівання потребують процесингу. Найкраще цей процес вивчений у плодової мухи Drosophila melanogaster. Первинних шлях процесингу піРНК не до кінця з'ясований, виконується, скоріше за все, за допомогою нуклеази Zucchini (Zuc), після розрізання такий прекурсор піРНК взаємодіє з Piwi чи Aubergine (Aub). У цьому процесі ще беруть участь білок теплового шоку 83 (Hsp83) та Shutdown (Shu). У результаті формується зріла піРНК, антисенсна транскрипту активного транспозону. Така піРНК вступає в «Пінг-понг» цикл, у якому вона з'єднується із транскриптом транспозону (сенс) і за допомогою Piwi чи Aubergine (Aub) розрізає його на зрілу піРНК (сенс). Потім цикл продовжується коли сенс піРНК та білок сімейства Аргонавт зв'язуються з антисенсним транскриптом з піРНК кластерів і розрізають його до довжини зрілої піРНК (антисенс). «Пінг-понг» цикл повторюється (див. ілюстрацію «Біогенез коротких некодуючих РНК»)[44].
Довгі некодуючі РНК[ред. | ред. код]

Довгі некодуючі РНК, днРНК (англ. lncRNA) — це великий клас РНК, що характеризується довжиною більше за 200 нуклеотидів та відсутністю відкритої рамки зчитування в їх послідовності, тобто їхня нуклеотидна послідовність не є кодом для амінокислотної послідовності білків.
Довгі некодуючі РНК мають багато спільних рис із мРНК, хоча їхній біогенез не так добре вивчений: вони часто синтезуються за допомогою РНК-полімерази II, поліаденілуються та проходять сплайсинг і навіть альтернативний сплайсинг[52]. При цьому деякі специфічні днРНК які знаходяться довгий час у ядрі, проте не мають класичного поліаденілування, зазвичай мають інші структури, які забезпечують стабільність. Наприклад, днРНК MALAT1[en] має цікаву три-ланцюгову структуру де і 5' і 3'-кінцеві ділянки знаходяться всередині і не підлягають нуклеазній деградації.[53]
ДнРНК можуть бути закодовані в геномі як у сенс- так і в антисенс-напрямках відносно генів, що кодують білки, можуть знаходитися в інтронах генів або бути міжгенними[54].
На 2014 рік ідентифіковано понад 10000 міжгенних довгих некодуючих РНК і багато інтронних. Довгі некодуючі РНК експресуються на нижчих рівнях, ніж протеїн-кодуючі РНК (мРНК), також вони часто є тканино-специфічними[55]. Функції днРНК слабко вивчені, але деякі з них регулюють рівні транскрипції певних генів шляхом безпосереднього зв'язування з факторами транскрипції, або завдяки епігенетичним механізмам регуляції експресії генів[56].
Довгі некодуючі РНК піддаються нуклеотидним модифікаціям, таким як метилювання цитозину та аденіну. Багато із цих посттранскрипційних модифікацій є оберненими, і скоріше за все, регулюють функції днРНК[57].
Здатність довгих некодуючих РНК згортатися у вторинну та третинну структури є основною характерною рисою функціонування цього класу некодуючих РНК. У структурі днРНК є шпильки, частини повної та неповної комплементарності, псевдовузли, що призводять до формування певної 3D моделі зі спіралями, які знаходяться паралельно чи перпендикулярно одна до одної — структури, які певним чином аналогічні елементам вторинної структури білків. Такі структури, вважається, і надають довгим некодуючим РНК можливість виконувати свої функції[57].
Редагування та модифікації РНК[ред. | ред. код]
Редагування РНК[ред. | ред. код]

Редагування РНК — це процес, при якому окремі нуклеотиди замінюються на інші в молекулі РНК. Також до редагування РНК відносять вставки та вирізання нуклеотидів РНК, що не є результатом сплайсингу. Редагування РНК змінює інформацію, закодовану в молекулі РНК і якщо це відбувається в кодуючій ділянці матричної РНК, то білок, що з неї буде зчитуватися, буде містити іншу амінокислоту. У тому випадку якщо додається/відрізається нуклеотид, — буде відбуватися зсув рамки зчитування[en], що призведе до кардинальної зміни амінокислот білка або деградація мРНК[58].
У процесі редагування РНК часто беруть участь багато різних білків та іноді також некодуючих РНК[58].
Редагування РНК присутнє у багатьох різних організмів. У рослин цей процес відбувається в мітохондріях і пластидах. У пластидах квіткових рослин від 30 до 40 цитозинів змінюються на урацили, тоді як у папоротей та мохоподібних ця цифра може досягати декількох сотень. Мітохондріальна ДНК квіткових рослин змінює приблизно 450 цитозинів на урацили, і в основному цей процес стосується мРНК, але у нижчих рослин Ц-У редагування РНК відбувається частіше (до 2000 нуклеотидів) і може проходити у зворотному напрямку[58].
У людини найчастіше відбувається зміна аденозину на інозин (A-на-I), що відбувається за допомогою сім'ї ферментів Аденозин-дезамінази РНК (англ. ADAR). ADAR здатні до з'єднання із дволанцюговими молекулами РНК і дезамінувати (прибрати аміно-групу, -NH2) аденозин до інозину. Інозин розшифровується іншими ферментами в основному як гуанозин. A-на-I редагування РНК часто відбувається в транспозонах, таких як Alu-повтори, тому що вони здатні формувати багато дволанцюгових РНК, тоді як було зафіксовано лише декілька десятків випадків редагування РНК в неповторювальних елементах геному (таких, як гени, що кодують білки), і більшість із них стосуються тканин нервової системи[59].
Іншим видом редагування РНК у людини є зміна Ц-на-У, яка виконується за допомогою інших дезаміназ, APOBEC. Але такий вид редагування РНК не є розповсюдженим і відбувається переважно в ентероцитах тонкого кишечнику[60] та в деяких інших клітинах (моноцити).
Редагування РНК є додатковим механізмом збільшення різноманіття РНК, а також способом контролю їхнього рівня, адже редагування може призводити до деградації молекули РНК.
Модифікації РНК[ред. | ред. код]
Під час дозрівання РНК різні ферменти можуть хімічно змінювати рибонуклеотиди[en]. Такі зміни можуть відбуватися як в азотистих основах, так і в 2' положенні рибози, чи одночасно і там і там. Також є модифікації, які відбуваються у декілька етапів поза молекулою РНК, а потім приєднуються до РНК за допомогою реакції нуклеотидної заміни. Прикладом такої реакції є кепування в деяких вірусів — до мРНК приєднується вже метильований гуанозин, метилювання якого пройшло на молекулі ГТФ[61]. На сьогодні відомо понад 100 різних хімічних модифікацій РНК, хоча функції більшості з них залишаються невідомими[62].
Оскільки РНК молекули, як вважалося, порівняно не довго існують у клітині, на сьогодні переважає думка, що модифікації рибонуклеотидів не довговічні, і після ковалентного з'єднання хімічної групи вона вже не від'єднується. Але є деякі відомості починаючи із 2011 року про обернене метилювання аденозину РНК (m6A)[62].
Найбільш редагованими з видів РНК є транспортні РНК — приблизно один із п'яти нуклеотидів тРНК є модифікованим, також відомо більше 50 різних видів модифікацій нуклеотидів тРНК.[63] Цікавим є те, що антикодонова петля є мішенню багатьох модифікацій нуклеотидів, при чому це залежить від того, яку амінокислоту і відповідно який антикодон містить дана тРНК. Оскільки послідовності антикодонових нуклеотидів різні, різними бувають модифікації тих нуклеотидів, які оточують антикодон (34-ий і 37-ий нуклеотиди найчастіше модифікуються), але вони забезпечують структурну відкритість антикодону і збільшують кодон-антикодонове впізнання.[63]
Модифікації уридину[ред. | ред. код]

- Псевдоуридин (Ψ)
Модифікація РНК, яка зустрічається найчастіше, це ізомеризація уридину на псевдоуридин (Ψ). Псевдоуридин, на відміну від урідину, здатен формувати додатковий водневий зв'язок, тому така модифікація призводить до збільшення структурної стабільності молекули РНК: пара U-A легше розплітається, ніж пара Ψ-A[24]. Так, у дріжджів при тепловому шоці більшість молекул РНК деградують за допомогою екзосомального комплексу, проте ті, які мають на своєму 3'-кінці псевдоуридин гірше плавляться і є більш стабільні, оскільки екзосомі треба мати вільний, одноланцюговий 3'-кінець для нуклеазної активності.[24]
У дріжджів псевдоуридин наявний у 46 позицій чотирьох рРНК (25S, 18S, 5.8S, та 5S), та у шести позиціях у малих ядерних РНК U1[en], U2[en], та U5[en]. Транспортна РНК отримує перетворений уридин у псевдоуридиновій петлі за допомогою спеціальних ферментів, псевдоуридин синтаз (англ. pseudouridine synthase, PUS). Людини має 23 білки з доменом псевдоуридин синтази, але вони не вивчені до кінця. У вересні 2014 року Schwartz та співавтори випустили у журналі Cell статтю про наявність псевдоуридину в молекулах мРНК та малих ядерних РНК як дріжджів так і людини та запропонували методику секвенування Ψ-Seq для виявлення псевдоуридинів у цілих транскриптомах[64].
Модифікації аденозину[ред. | ред. код]

|

|
|
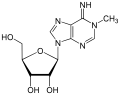
| Аденозин | N6-метиладенозин | N1-метиладенозин |
- N6-метиладенозин (m6A)
Дана модифікація є найбільш розповсюдженою з усіх модифікацій мРНК еукаріот і вона становить приблизно 80 % змінених нуклеотидів мРНК.[24]
Модифікація m6A зустрічається в 3'-нетрансльованій ділянці мРНК. Експериментальне виключення ферментів, що додають m6A, призводить до порушення сплайсингу сотень генів, що дає підстави вважати що дана модифікація впливає на сплайсинг.[24]
Присутність одного модифікованого m6A в 5'-нетрансльованій ділянці дозволяє запустити кеп-незалежну транскрипцію мРНК теплового шоку HSP70, тоді як кеп-залежна транскрипція заглушується під час теплового шоку.[24]
Також m6A зустрічається в інтронах.[24]
- N1-метиладенозин (m1A)
N1-метиладенозин вносить позитивний заряд у Вотсон-Криківську взаємодію, таким чином дана модифікація може сильно змінити вторинну структуру РНК чи взаємодію РНК з білками[63]
Більшість транскриптів, які мають N1-метиладенозин, містять лише один сайт m1A в 5'-нетрансльованій ділянці. Такі мРНК мають зазвичай білшу стабільність і вищій рівень трансляції.[24]
Модифікації цитозину[ред. | ред. код]

|
|

|
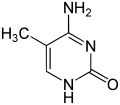
| Цитозин | 5-Метилцитозин | 5-Гідроксиметилцитозин |
- 5-метилцитозин (m5C) та 5-гідроксиметилцитозин (hm5C)
5-Метилцитозин бере участь у регуляції трансляції, залежно від того, в якій частині мРНК розташований цей модифікований нуклеотид. Так, присутність m5C у 3'-нетрансльованому регіоні CDK1 мРНК збільшує рівень трансляції, а у 5'-НТР мРНК CDKN1B навпаки знижує. Такий механізм допомагає регулювати клітинний цикл.[24]
Перетворення 5-метилцитозину на 5-гідроксиметилцитозин відбувається за допомогою ферментів метилцитозин-деоксигеназ. Така модифікація в плодових мух корилює із присутністю полірибосом.[24]
Модифікації рибози[ред. | ред. код]

|

|
| Уридин | 2′-O-метилуридин |
- 2′-OMe
Основною вивченою модифікацією цукру РНК є метилювання у 2′-позиції залишку рибози, 2′-OMe[en]: OH-група у положенні 2'C замінюється на OCH3. Така модифікація загалом збільшує стабільність структури РНК[65]
Порушення процесингу РНК та хвороби[ред. | ред. код]
Нормальне функціонування клітин залежить від суворого контролю рівня експресії як РНК, що кодують білки, так і некодуючих РНК. Такі РНК беруть участь у транскрипції, процесингу та трансляції, підтриманні довжини теломер та багатьох інших подіях у клітині. Оскільки процесинг РНК включає в себе дозрівання молекули РНК від тієї форми, що закодована в молекулі ДНК, до зрілої функціональної РНК, то порушення цього процесу може викликати захворювання.
Так, при виникненні ізоформ мРНК — наприклад, у результаті мутацій, які призводять до активації іншого сайту сплайсингу — білки, які зчитуються з таких матриць можуть мати інший амінокислотний склад або бути конформаційно нестабільними, що призводить до нездатності білка виконувати свої функції[66]. Прикладів альтернативного сплайсингу, який призводить до захворювань, є безліч. Так при атаксії телеангіектазії (синдром Луї-Бар), нейродегенеративному захворюванні зі схильністю до злоякісних новоутворень, делеція 4 нуклеотидів у 20-му інтроні гена ATM (англ. ataxia-telengiectasia mutated) призводить до активації альтернативного сплайсингу, та спричинює розвиток захворювання[67].
РНК існують у клітинах у зв'язаному з білками стані, у вигляді так званих рибонуклеопротеїнових комплексів (РНП, англ. RNP) що складаються з однієї або більше молекул РНК та найчастіше багатьох РНК-зв'язуючих білків[en] (англ. RNA-binding proteins, RBP, RNABP)[68]. Власне, виконання відповідними РНК своїх функцій відбувається в таких рибонуклеопротеїнових комплексах, і їх нормальна активність залежить від чіткого розташування білкових структур відносно третинної структури РНК. Збої під час процесингу як відповідних некодуючих РНК, так і мРНК, що кодують ці білки, можуть призвести до порушення утворення цих комплексів[68]. Наприклад, РНК-зв'язуючі білки, що в нормальних умовах беруть участь у регуляції сплайсингу, формують нетипові агрегати при хворобі Паркінсона та при аміотрофічному бічному склерозі[11].
Цікавим випадком є синонімічні мутації — такі мутації в гені, що припадають на кодуючу ділянку РНК і не призводять до зміни амінокислоти, що вони кодують. Наприклад, ГГТ, ГГА та ГГГ кодують одну амінокислоту — гліцин. При точковій мутації гена в третьому положені цього кодону ГГ_ (наприклад ГГА→ГГЦ), амінокислота, що кодується такою мРНК, не зміниться — це все одно буде гліцин, звідси і назва мутації — синонімічна, адже в даному випадку А синонімічний Ц. Довгий час вважалося, що синонімічні мутації не призводять до будь-якого впливу на функціонування клітини. Однак у деяких випадках до 25 % таких синонімічних мутацій можуть впливати на взаємодію зі сплайсосомою і призводити до альтернативного сплайсингу[68].
Див. також[ред. | ред. код]
- Експресія генів
- Деградація РНК
- Епігенетика
- Епігенетичний код
- Регуляторні послідовності
- Оперон
- Антисенсові РНК
- Посттрансляційна модифікація
Примітки[ред. | ред. код]
- ↑ а б А. В. Сиволоб (2008). Молекулярна біологія (PDF). К: Видавничо-поліграфічний центр "Київський університет". с. а. 201–220 б. 207-208. Архів оригіналу (PDF) за 4 березня 2016. Процитовано 20 червня 2014.
- ↑ а б в г David L. Bentley (March 2014). Coupling mRNA processing with transcription in time and space. Nature reviews. Genetics. 15 (3): 163—175. doi:10.1038/nrg3662. PMID 24514444.
- ↑ Takayuki Ohira & Tsutomu Suzuki (June 2011). Retrograde nuclear import of tRNA precursors is required for modified base biogenesis in yeast. Proceedings of the National Academy of Sciences of the United States of America. 108 (26): 10502—10507. doi:10.1073/pnas.1105645108. PMID 21670254.
- ↑ а б в г Alberto R. Kornblihtt, Ignacio E. Schor, Mariano Allo, Gwendal Dujardin, Ezequiel Petrillo & Manuel J. Munoz (March 2013). Alternative splicing: a pivotal step between eukaryotic transcription and translation. Nature reviews. Molecular cell biology. 14 (3): 153—165. doi:10.1038/nrm3525. PMID 23385723.
- ↑ Diana Y. Vargas, Khyati Shah, Mona Batish, Michael Levandoski, Sourav Sinha, Salvatore A. E. Marras, Paul Schedl & Sanjay Tyagi (November 2011). Single-molecule imaging of transcriptionally coupled and uncoupled splicing. Cell. 147 (5): 1054—1065. doi:10.1016/j.cell.2011.10.024. PMID 22118462.
- ↑ Schmid, Manfred; Jensen, Torben Heick (08 2018). Controlling nuclear RNA levels. Nature Reviews. Genetics. Т. 19, № 8. с. 518—529. doi:10.1038/s41576-018-0013-2. ISSN 1471-0064. PMID 29748575.
- ↑ Zlotorynski, Eytan (2016). RNA metabolism: Co-transcriptional splicing at nucleotide resolution. Nature Reviews Molecular Cell Biology. 17 (5): 264—265. doi:10.1038/nrm.2016.44. ISSN 1471-0072.
- ↑ Michael Hiller, Klaus Huse, Matthias Platzer & Rolf Backofen (2005). Creation and disruption of protein features by alternative splicing -- a novel mechanism to modulate function. Genome biology. 6 (7): R58. doi:10.1186/gb-2005-6-7-r58. PMID 15998447.
{{cite journal}}: Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання) - ↑ Bradley E. Bernstein, Ewan Birney, Ian Dunham, Eric D. Green, Chris Gunter & Michael Snyder (September 2012). An integrated encyclopedia of DNA elements in the human genome. Nature. 489 (7414): 57—74. doi:10.1038/nature11247. PMID 22955616.
- ↑ Qun Pan, Ofer Shai, Leo J. Lee, Brendan J. Frey & Benjamin J. Blencowe (December 2008). Deep surveying of alternative splicing complexity in the human transcriptome by high-throughput sequencing. Nature genetics. 40 (12): 1413—1415. doi:10.1038/ng.259. PMID 18978789.
- ↑ а б в Xiang-Dong Fu & Manuel Jr Ares (August 2014). Context-dependent control of alternative splicing by RNA-binding proteins. Nature reviews. Genetics. doi:10.1038/nrg3778. PMID 25112293.
- ↑ а б в Baralle, Francisco E.; Giudice, Jimena (July 2017). Alternative splicing as a regulator of development and tissue identity. Nature Reviews. Molecular Cell Biology. Т. 18, № 7. с. 437—451. doi:10.1038/nrm.2017.27. ISSN 1471-0080. PMID 28488700. Архів оригіналу за 26 січня 2018. Процитовано 17 жовтня 2017.
- ↑ Білок NOVA-1 людини в датабазі UniProt P51513
- ↑ Білок PTB людини в базі даних UniProt P26599
- ↑ Kian Huat Lim, Luciana Ferraris, Madeleine E. Filloux, Benjamin J. Raphael & William G. Fairbrother (July 2011). Using positional distribution to identify splicing elements and predict pre-mRNA processing defects in human genes. Proceedings of the National Academy of Sciences of the United States of America. 108 (27): 11093—11098. doi:10.1073/pnas.1101135108. PMID 21685335.
- ↑ а б в Ran Elkon, Alejandro P. Ugalde & Reuven Agami (July 2013). Alternative cleavage and polyadenylation: extent, regulation and function. Nature reviews. Genetics. 14 (7): 496—506. doi:10.1038/nrg3482. PMID 23774734.
- ↑ а б Laure Weill, Eulalia Belloc, Felice-Alessio Bava & Raul Mendez (June 2012). Translational control by changes in poly(A) tail length: recycling mRNAs. Nature structural & molecular biology. 19 (6): 577—585. doi:10.1038/nsmb.2311. PMID 22664985.
- ↑ а б в г д Dafne Campigli Di Giammartino, Kensei Nishida & James L. Manley (September 2011). Mechanisms and consequences of alternative polyadenylation. Molecular cell. 43 (6): 853—866. doi:10.1016/j.molcel.2011.08.017. PMID 21925375.
- ↑ Susan Carpenter, Emiliano P. Ricci, Blandine C. Mercier, Melissa J. Moore & Katherine A. Fitzgerald (June 2014). Post-transcriptional regulation of gene expression in innate immunity. Nature reviews. Immunology. 14 (6): 361—376. doi:10.1038/nri3682. PMID 24854588.
- ↑ Katarzyna Oktaba, Wei Zhang, Thea Sabrina Lotz, David Jayhyun Jun, Sandra Beatrice Lemke, Samuel Pak Ng, Emilia Esposito, Michael Levine & Valerie Hilgers (December 2014). ELAV Links Paused Pol II to Alternative Polyadenylation in the Drosophila Nervous System. Molecular cell. doi:10.1016/j.molcel.2014.11.024. PMID 25544561.
- ↑ Zlotorynski Eytan (2015). RNA metabolism: Promoters drive alternative polyadenylation. Nature Reviews Molecular Cell Biology. 16 (2): 66—66. doi:10.1038/nrm3940. ISSN 1471-0072.
- ↑ Zlotorynski Eytan (2015). RNA: 3′ UTR alternatives to protein localization. Nature Reviews Molecular Cell Biology. 16 (6): 327—327. doi:10.1038/nrm3996. ISSN 1471-0072.
- ↑ Yang, Li; Duff, Michael O.; Graveley, Brenton R.; Carmichael, Gordon G.; Chen, Ling-Ling (16 лютого 2011). Genomewide characterization of non-polyadenylated RNAs. Genome Biology. Т. 12. с. R16. doi:10.1186/gb-2011-12-2-r16. ISSN 1474-760X.
{{cite news}}: Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання) - ↑ а б в г д е ж и к л Lewis, Cole J. T.; Pan, Tao; Kalsotra, Auinash (March 2017). RNA modifications and structures cooperate to guide RNA-protein interactions. Nature Reviews. Molecular Cell Biology. Т. 18, № 3. с. 202—210. doi:10.1038/nrm.2016.163. ISSN 1471-0080. PMC 5542016. PMID 28144031. Архів оригіналу за 26 січня 2018. Процитовано 11 жовтня 2017.
{{cite news}}: Cite має пустий невідомий параметр:|18=(довідка)Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання) - ↑ а б в г д William F. Marzluff, Eric J. Wagner & Robert J. Duronio (November 2008). Metabolism and regulation of canonical histone mRNAs: life without a poly(A) tail. Nature reviews. Genetics. 9 (11): 843—854. doi:10.1038/nrg2438. PMID 18927579.
- ↑ Білок ZFP100 людини UniProt Q8WTR7
- ↑ Білок LSM11 людини UniProt P83369
- ↑ Білок LSM10 людини UniProt Q969L4
- ↑ А. В. Сиволоб, С.Р. Рушковський, С.С. Кир'яченко та ін. (2008). Генетика (PDF). К: Видавничо-поліграфічний центр "Київський університет". с. 57—58. Архів оригіналу (PDF) за 4 березня 2016. Процитовано 16 липня 2014.
{{cite book}}: Явне використання «та ін.» у:|author=(довідка) - ↑ а б Anita K. Hopper & Eric M. Phizicky (January 2003). tRNA transfers to the limelight. Genes & development. 17 (2): 162—180. doi:10.1101/gad.1049103. PMID 12533506.
- ↑ Liande Li, Weifeng Gu, Chunyang Liang, Qinghua Liu, Craig C. Mello & Yi Liu (August 2012). The translin-TRAX complex (C3PO) is a ribonuclease in tRNA processing. Nature structural & molecular biology. 19 (8): 824—830. doi:10.1038/nsmb.2337. PMID 22773104.
- ↑ Toshikatsu Hanada, Stefan Weitzer, Barbara Mair, Christian Bernreuther, Brian J. Wainger, Justin Ichida та ін. (March 2013). CLP1 links tRNA metabolism to progressive motor-neuron loss. Nature. 495 (7442): 474—480. doi:10.1038/nature11923. PMID 23474986.
{{cite journal}}: Явне використання «та ін.» у:|author=(довідка) - ↑ Birgit S. Budde, Yasmin Namavar, Peter G. Barth та ін. (September 2008). tRNA splicing endonuclease mutations cause pontocerebellar hypoplasia. Nature genetics. 40 (9): 1113—1118. doi:10.1038/ng.204. PMID 18711368.
{{cite journal}}: Явне використання «та ін.» у:|author=(довідка) - ↑ Christopher R. Trotta, Sergey V. Paushkin, Meenal Patel, Hong Li & Stuart W. Peltz (May 2006). Cleavage of pre-tRNAs by the splicing endonuclease requires a composite active site. Nature. 441 (7091): 375—377. doi:10.1038/nature04741. PMID 16710424.
- ↑ Markus Englert & Hildburg Beier (2005). Plant tRNA ligases are multifunctional enzymes that have diverged in sequence and substrate specificity from RNA ligases of other phylogenetic origins. Nucleic acids research. 33 (1): 388—399. doi:10.1093/nar/gki174. PMID 15653639.
- ↑ Akiko Soma, Junichi Sugahara, Akinori Onodera, Nozomu Yachie та ін. (2013). Identification of highly-disrupted tRNA genes in nuclear genome of the red alga, Cyanidioschyzon merolae 10D. Scientific reports. 3: 2321. doi:10.1038/srep02321. PMID 23900518.
{{cite journal}}: Явне використання «та ін.» у:|author=(довідка) - ↑ а б Francois-Michel Boisvert, Silvana van Koningsbruggen, Joaquin Navascues & Angus I. Lamond (July 2007). The multifunctional nucleolus. Nature reviews. Molecular cell biology. 8 (7): 574—585. doi:10.1038/nrm2184. PMID 17519961.
- ↑ Jan van Riggelen, Alper Yetil & Dean W. Felsher (April 2010). MYC as a regulator of ribosome biogenesis and protein synthesis. Nature reviews. Cancer. 10 (4): 301—309. doi:10.1038/nrc2819. PMID 20332779.
- ↑ D. L. Lafontaine & D. Tollervey (July 2001). The function and synthesis of ribosomes. Nature reviews. Molecular cell biology. 2 (7): 514—520. doi:10.1038/35080045. PMID 11433365.
- ↑ Martin Ciganda & Noreen Williams (July-August 2011). Eukaryotic 5S rRNA biogenesis. Wiley interdisciplinary reviews. RNA. 2 (4): 523—533. doi:10.1002/wrna.74. PMID 21957041.
- ↑ а б W. S. Vincent Yip, Nicholas G. Vincent & Susan J. Baserga (2013). Ribonucleoproteins in archaeal pre-rRNA processing and modification. Archaea. 2013: 614735. doi:10.1155/2013/614735. PMID 23554567.
{{cite journal}}: Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання) - ↑ Taiping Chen & Sharon Y. R. Dent (February 2014). Chromatin modifiers and remodellers: regulators of cellular differentiation. Nature reviews. Genetics. 15 (2): 93—106. doi:10.1038/nrg3607. PMID 24366184.
- ↑ Deborah Bourc'his & Olivier Voinnet (October 2010). A small-RNA perspective on gametogenesis, fertilization, and early zygotic development. Science. 330 (6004): 617—622. doi:10.1126/science.1194776. PMID 21030645.
- ↑ а б Maartje J. Luteijn & Rene F. Ketting (August 2013). PIWI-interacting RNAs: from generation to transgenerational epigenetics. Nature reviews. Genetics. 14 (8): 523—534. doi:10.1038/nrg3495. PMID 23797853.
- ↑ Irfan A. Qureshi & Mark F. Mehler (August 2012). Emerging roles of non-coding RNAs in brain evolution, development, plasticity and disease. Nature reviews. Neuroscience. 13 (8): 528—541. doi:10.1038/nrn3234. PMID 22814587.
- ↑ а б Stephane E. Castel & Robert A. Martienssen (February 2013). RNA interference in the nucleus: roles for small RNAs in transcription, epigenetics and beyond. Nature reviews. Genetics. 14 (2): 100—112. doi:10.1038/nrg3355. PMID 23329111.
- ↑ Eleanor White, Margarita Schlackow, Kinga Kamieniarz-Gdula, Nick J. Proudfoot & Monika Gullerova (June 2014). Human nuclear Dicer restricts the deleterious accumulation of endogenous double-stranded RNA. Nature structural & molecular biology. 21 (6): 552—559. doi:10.1038/nsmb.2827. PMID 24814348.
- ↑ а б Eugene Berezikov (December 2011). Evolution of microRNA diversity and regulation in animals. Nature reviews. Genetics. 12 (12): 846—860. doi:10.1038/nrg3079. PMID 22094948.
- ↑ Susanne Rother & Gunter Meister (November 2011). Small RNAs derived from longer non-coding RNAs. Biochimie. 93 (11): 1905—1915. doi:10.1016/j.biochi.2011.07.032. PMID 21843590.
- ↑ Amy E. Pasquinelli (April 2012). MicroRNAs and their targets: recognition, regulation and an emerging reciprocal relationship. Nature reviews. Genetics. 13 (4): 271—282. doi:10.1038/nrg3162. PMID 22411466.
- ↑ а б в Benjamin Czech & Gregory J. Hannon (January 2011). Small RNA sorting: matchmaking for Argonautes. Nature reviews. Genetics. 12 (1): 19—31. doi:10.1038/nrg2916. PMID 21116305.
- ↑ а б в Kevin V. Morris & John S. Mattick (June 2014). The rise of regulatory RNA. Nature reviews. Genetics. 15 (6): 423—437. doi:10.1038/nrg3722. PMID 24776770.
- ↑ Brown, Jessica A.; Bulkley, David; Wang, Jimin; Valenstein, Max L.; Yario, Therese A.; Steitz, Thomas A.; Steitz, Joan A. (2014-7). Structural insights into the stabilization of MALAT1 noncoding RNA by a bipartite triple helix. Nature Structural & Molecular Biology. Т. 21, № 7. с. 633—640. doi:10.1038/nsmb.2844. ISSN 1545-9985. PMC 4096706. PMID 24952594. Архів оригіналу за 12 листопада 2018. Процитовано 20 листопада 2019.
- ↑ S. W. Cheetham, F. Gruhl, J. S. Mattick & M. E. Dinger (June 2013). Long noncoding RNAs and the genetics of cancer. British journal of cancer. 108 (12): 2419—2425. doi:10.1038/bjc.2013.233. PMID 23660942.
- ↑ Keith W. Vance & Chris P. Ponting (August 2014). Transcriptional regulatory functions of nuclear long noncoding RNAs. Trends in genetics : TIG. 30 (8): 348—355. doi:10.1016/j.tig.2014.06.001. PMID 24974018.
- ↑ Martin Turner, Alison Galloway & Elena Vigorito (June 2014). Noncoding RNA and its associated proteins as regulatory elements of the immune system. Nature immunology. 15 (6): 484—491. doi:10.1038/ni.2887. PMID 24840979.
- ↑ а б Tim R. Mercer & John S. Mattick (March 2013). Structure and function of long noncoding RNAs in epigenetic regulation. Nature structural & molecular biology. 20 (3): 300—307. doi:10.1038/nsmb.2480. PMID 23463315.
- ↑ а б в Mizuki Takenaka & Axel Brennicke (November 2012). Using multiplex single-base extension typing to screen for mutants defective in RNA editing. Nature protocols. 7 (11): 1931—1945. doi:10.1038/nprot.2012.117. PMID 23037308.
- ↑ Gokul Ramaswami, Rui Zhang, Robert Piskol, Liam P. Keegan, Patricia Deng, Mary A. O'Connell & Jin Billy Li (February 2013). Identifying RNA editing sites using RNA sequencing data alone. Nature methods. 10 (2): 128—132. doi:10.1038/nmeth.2330. PMID 23291724.
- ↑ Gokul Ramaswami, Wei Lin, Robert Piskol, Meng How Tan, Carrie Davis & Jin Billy Li (June 2012). Accurate identification of human Alu and non-Alu RNA editing sites. Nature methods. 9 (6): 579—581. doi:10.1038/nmeth.1982. PMID 22484847.
- ↑ Magdalena A. Machnicka, Kaja Milanowska, Okan Osman Oglou, Elzbieta Purta та ін. (January 2013). MODOMICS: a database of RNA modification pathways--2013 update. Nucleic acids research. 41 (Database issue): D262—D267. doi:10.1093/nar/gks1007. PMID 23118484.
{{cite journal}}: Явне використання «та ін.» у:|author=(довідка) - ↑ а б Ye Fu, Dan Dominissini, Gideon Rechavi & Chuan He (May 2014). Gene expression regulation mediated through reversible m(6)A RNA methylation. Nature reviews. Genetics. 15 (5): 293—306. doi:10.1038/nrg3724. PMID 24662220.
- ↑ а б в Roundtree, Ian A.; Evans, Molly E.; Pan, Tao; He, Chuan (15 червня 2017). Dynamic RNA Modifications in Gene Expression Regulation. Cell. Т. 169, № 7. с. 1187—1200. doi:10.1016/j.cell.2017.05.045. ISSN 1097-4172. PMID 28622506. Архів оригіналу за 26 січня 2018. Процитовано 14 жовтня 2017.
{{cite news}}: Cite має пустий невідомий параметр:|19=(довідка) - ↑ Schwartz Schraga, Bernstein Douglas A., Mumbach Maxwell R. та ін. (September 2014). Transcriptome-wide Mapping Reveals Widespread Dynamic-Regulated Pseudouridylation of ncRNA and mRNA. Cell. doi:10.1016/j.cell.2014.08.028. ISSN 0092-8674.
{{cite journal}}: Явне використання «та ін.» у:|author=(довідка) - ↑ Yildirim, Ilyas; Kierzek, Elzbieta; Kierzek, Ryszard; Schatz, George C. (11 грудня 2014). Interplay of LNA and 2'-O-methyl RNA in the structure and thermodynamics of RNA hybrid systems: a molecular dynamics study using the revised AMBER force field and comparison with experimental results. The Journal of Physical Chemistry. B. Т. 118, № 49. с. 14177—14187. doi:10.1021/jp506703g. ISSN 1520-5207. PMID 25268896. Архів оригіналу за 3 листопада 2017. Процитовано 20 жовтня 2017.
{{cite news}}: Cite має пустий невідомий параметр:|19=(довідка) - ↑ Donny D. Licatalosi & Robert B. Darnell (January 2010). RNA processing and its regulation: global insights into biological networks. Nature reviews. Genetics. 11 (1): 75—87. doi:10.1038/nrg2673. PMID 20019688.
- ↑ Donny D. Licatalosi & Robert B. Darnell (October 2006). Splicing regulation in neurologic disease. Neuron. 52 (1): 93—101. doi:10.1016/j.neuron.2006.09.017. PMID 17015229.
- ↑ а б в Thomas A. Cooper, Lili Wan & Gideon Dreyfuss (February 2009). RNA and disease. Cell. 136 (4): 777—793. doi:10.1016/j.cell.2009.02.011. PMID 19239895.
Джерела[ред. | ред. код]
- David L. Bentley (March 2014). Coupling mRNA processing with transcription in time and space. Nature reviews. Genetics. 15 (3): 163—175. doi:10.1038/nrg3662. PMID 24514444.
- А. В. Сиволоб (2008). Молекулярна біологія (PDF). К: Видавничо-поліграфічний центр "Київський університет". Архів оригіналу (PDF) за 4 березня 2016. Процитовано 20 червня 2014.
- А. В. Сиволоб, С.Р. Рушковський, С.С. Кир'яченко та ін. (2008). Генетика (PDF). К: Видавничо-поліграфічний центр "Київський університет". Архів оригіналу (PDF) за 4 березня 2016. Процитовано 16 липня 2014.
{{cite book}}: Явне використання «та ін.» у:|author=(довідка)
| ||||||||||||||||||||||||||||||||||||||||||||||
|
|
Ця стаття належить до вибраних статей Української Вікіпедії. |
|
|
Ця стаття належить до добрих статей української Вікіпедії. |

