Конденсин

Конденсин — великий білковий комплекс, який відіграє центральну роль у конденсації та сегрегації хромосом під час мітозу та мейозу.[1][2]
Субодиниці[ред. | ред. код]
Евкаріотичні конденсини[ред. | ред. код]

У багатьох евкаріотичних клітинах комплекси конденсину представлені двома типами: конденсин I та конденсин II, кожен з яких складається з п'яти субодиниць.[3][4] Конденсини I та II мають однакову пару ключових субодиниць, SMC2 та SMC4, що належать до великої групи хромосомних АТФаз, відомих як SMC-білки (SMC від Structural Maintenance of Chromosomes). Кожен з комплексів конденсину також містить набір не-SMC-регуляторних субодиниць (клейзин[5] а також пару HEAT-повтор субодиниць[уточнити][6]). Нематода Caenorhabditis elegans має також третій тип комплексу (схожий на конденсин I) який бере участь у регуляції експресії генів, на рівні хромосом, тобто у дозовій компенсації.[7] В цьому комплексі, відомому як конденсин IDC, аутентична субодиниця SMC4 замінена на її варіант DPY-27.
| Комплекс | Субодиниця | Класифікація | S. cerevisiae | S. pombe | C. elegans | D. melanogaster | Хребетні (людина) |
|---|---|---|---|---|---|---|---|
| конденсин I & II | SMC2 | ATPase | Smc2 | Cut14 | MIX-1 | DmSmc2 | CAP-E (SMC2) |
| конденсин I & II | SMC4 | ATPase | Smc4 | Cut3 | SMC-4 | DmSmc4 | CAP-C (SMC4) |
| конденсин I | CAP-D2 | HEAT | Ycs4 | Cnd1 | DPY-28 | CG1911 | CAP-D2 (NCAPD2) |
| конденсин I | CAP-G | HEAT | Ycg1 | Cnd3 | CAP-G1 | cap-g | CAP-G (NCAPG) |
| конденсин I | CAP-H | kleisin | Brn1 | Cnd2 | DPY-26 | barren | CAP-H (NCAPH) |
| конденсин II | CAP-D3 | HEAT | - | - | HCP-6 | CG31989 | CAP-D3 (NCAPD3) |
| конденсин II | CAP-G2 | HEAT | - | - | CAP-G2 | -? | CAP-G2 (NCAPG2) |
| конденсин II | CAP-H2 | kleisin | - | - | KLE-2 | CG14685 | CAP-H2 (NCAPH2) |
| конденсин IDC | SMC4 варіант | ATPase | - | - | DPY-27 | - | - |
Структура та функція конденсину I є консервативними від дріжджів до людини, хоча при цьому дріжджі не мають конденсину II.[8][9] Немає очевидного зв'язку між появою конденсину II та розміром евкаріотичного геному. Наприклад, червона водорость Cyanidioschyzon merolae має обидва конденсини (I та II) хоча її геном малий та співрозмірний з геномом дріжджів.[10]
Прокаріотичний конденсин[ред. | ред. код]
Прокаріоти мають конденсиноподібні комплекси, які також залучені до організації та сегрегації хромосом. Прокаріотичні конденсини можуть бути розділені на дві групи: SMC-ScpAB[11] та MukBEF.[12] Багато бактерій та архей мають SMC-ScpAB, тоді як підгрупа, відома як гамма-протобактерії, мають MukBEF.
| Комплекс | Субодин. | класифікація | B. subtilis | Caulobacter | E.coli |
|---|---|---|---|---|---|
| SMC-ScpAB | SMC | ATPase | SMC/BsSMC | SMC | - |
| SMC-ScpAB | ScpA | kleisin | ScpA | ScpA | - |
| SMC-ScpAB | ScpB | winged-helix | ScpB | ScpB | - |
| MukBEF | MukB | ATPase | - | - | MukB |
| MukBEF | MukE | ? | - | - | MukE |
| MukBEF | MukF | kleisin | - | - | MukF |
Молекулярні механізми[ред. | ред. код]
Молекулярна активність[ред. | ред. код]
Очищений конденсин I створює позитивну суперспіралізацію в дволанцюговій ДНК у АТФ-гідроліз-залежний спосіб.[13] Він також володіє ДНК-стимульованою АТФазною активністю in vitro. Димер SMC2-SMC4 має здатність гібридизувати роз'єднані комплементарні одноланцюгові ДНК.[14] Ця активність не залежить від АТФ.
Молекулярна структура[ред. | ред. код]
Димер SMC-субодиниць має унікальну V-подібну форму.[15] Гомокомплекс конденсину І був візуалізований за допомогою електронної мікроскопії.[16]
Мітотичні функції[ред. | ред. код]
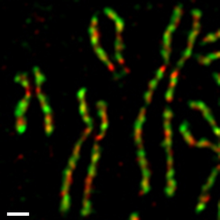
В культурі людських клітин обидва комплекси конденсину протягом клітинного циклу регулюються по-різному.[17][18] Конденсин II присутній в ядрі клітин під час інтерфази та бере участь у ранніх стадіях конденсації хромосом в профазі. Конденсин I локалізований в цитоплазмі під час інтерфази, і отримує доступ до хромосомної ДНК тільки після розчинення ядерної мембрани в пізній профазі–ранній прометафазі. Під час прометафази та метафази конденсин I та конденсин II беруть участь в утриманні хроматину в сконденсованому стані, при цьому сестринські хроматиди можна повністю розрізнити.[4] Обидва білка залишаються в цьому ж асоційованому стані коли хроматиди роз*єднуються і розходяться в анафазі. Як мінімум одна субодиниця конденсину I є прямою мішенню циклін-залежної кінази (Cdk).[19]
Роль в організації інтерфазних хромосом[ред. | ред. код]
Дослідження показали що конденсини беруть участь в різноманітних функціях по організації хромосом поза рамками мітозу. В дріжджах конденсин I (єдиний конденсин організму) бере участь у регуляції числа копій повторів рДНК[20] а також в кластеруванні генів тРНК.[21] В Drosophila субодиниці конденсину II беруть участь в дисоціації політенних хромосом[22] та в формуванні хромосомних територій[23] в деяких типах клітин.
Споріднені білки[ред. | ред. код]
Еукаріотичні клітини мають також два додаткових класи SMC-подібних комплексів. Когезин містить SMC1 та SMC3 та відповідає за когезію сестринських хроматид під час мітозу. Комплекс SMC5/6 містить білки SMC5 та SMC6 та залучений до рекомбінантної репарації ДНК.
Посилання[ред. | ред. код]
- ↑ Tatsuya Hirano. Condensins: universal organizers of chromosomes with diverse functions // Genes Dev.. — 2012. — Т. 26. — С. 1659–1678. — DOI:. Архівовано з джерела 10 грудня 2019. Процитовано.
- ↑ Wood AJ, Severson, AF, Meyer BJ. Condensin and cohesin complexity: the expanding repertoire of functions // Nat Rev Genet.. — 2010. — Т. 11, № 6. — С. 391–404. — DOI:. Архівовано з джерела 10 грудня 2019. Процитовано.
- ↑ Hirano T, Kobayashi R, Hirano M. Condensins, Chromosome Condensation Protein Complexes Containing XCAP-C, XCAP-E and a Xenopus Homolog of the Drosophila Barren Protein // Cell. — 1997. — Т. 89, № 4. — С. 511–521. — DOI:.
- ↑ а б Ono T, Losada A, Hirano M, Myers MP, Neuwald AF, Hirano T. Differential contributions of condensin I and condensin II to mitotic chromosome architecture in vertebrate cells // Cell. — 2003. — Т. 115, № 1. — С. 109–21. — DOI:.
- ↑ Schleiffer A, Kaitna S, Maurer-Stroh S, Glotzer M, Nasmyth K, Eisenhaber F. (2003). «Kleisins: a superfamily of bacterial and eukaryotic SMC protein partners». Mol. Cell 11 (3): 571–5. doi:10.1016/S1097-2765(03)00108-4. PMID 12667442
- ↑ Neuwald AF, Hirano T. (2000). «HEAT repeats associated with condensins, cohesins, and other complexes involved in chromosome-related functions». Genome Res. 10 (10): 1445–52. doi:10.1101/gr.147400. PMID 11042144
- ↑ Csankovszki G, Collette K, Spahl K, Carey J, Snyder M, Petty E, Patel U, Tabuchi T, Liu H, McLeod I, Thompson J, Sarkeshik A, Yates J, Meyer BJ, Hagstrom K. (2009). «Three distinct condensin complexes control C. elegans chromosome dynamics». Curr. Biol. 19 (1): 9–19. doi:10.1016/j.cub.2008.12.006. PMID 19119011
- ↑ Sutani T, Yuasa T, Tomonaga T, Dohmae N, Takio K, Yanagida M. (1999). «Fission yeast condensin complex: essential roles of non-SMC subunits for condensation and Cdc2 phosphorylation of Cut3/SMC4». Genes Dev. 13 (17): 2271–83. doi:10.1101/gad.13.17.2271. PMID 10485849
- ↑ Freeman L, Aragon-Alcaide L, Strunnikov A. (2000). «The condensin complex governs chromosome condensation and mitotic transmission of rDNA». J. Cell Biol. 149 (4): 811—824. doi:10.1083/jcb.149.4.811. PMID 10811823
- ↑ Fujiwara T, Tanaka K, Kuroiwa T, Hirano T. (2013). «Spatiotemporal dynamics of condensins I and II: evolutionary insights from the primitive red alga Cyanidioschyzon merolae». Mol. Biol. Cell. 24 (16): 2515–27. doi:10.1091/mbc.E13-04-0208. PMID 23783031
- ↑ Mascarenhas J, Soppa J, Strunnikov AV, Graumann PL. (2002). "Cell cycle-dependent localization of two novel prokaryotic chromosome segregation and condensation proteins in Bacillus subtilis that interact with SMC protein". EMBO J. 21 (12): 3108–18. doi:10.1093/emboj/cdf314. PMID 12065423
- ↑ Yamazoe M, Onogi T, Sunako Y, Niki H, Yamanaka K, Ichimura T, Hiraga S. (1999). "Complex formation of MukB, MukE and MukF proteins involved in chromosome partitioning in Escherichia coli". EMBO J. 18 (21): 5873–84. doi:10.1093/emboj/18.21.5873. PMID 10545099
- ↑ Kimura K, Hirano T. (1997). "ATP-dependent positive supercoiling of DNA by 13S condensin: a biochemical implication for chromosome condensation". Cell 90 (4): 625–634. doi:10.1016/s0092-8674(00)80524-3. PMID 9288743
- ↑ Sutani T, Yanagida M. (1997). "DNA renaturation activity of the SMC complex implicated in chromosome condensation". Nature 388 (6644): 798–801. doi:10.1038/42062. PMID 9285594
- ↑ Melby TE, Ciampaglio CN, Briscoe G, Erickson HP. (1998). «The symmetrical structure of structural maintenance of chromosomes (SMC) and MukB proteins: long, antiparallel coiled coils, folded at a flexible hinge». J. Cell Biol. 142 (6): 1595—1604. doi:10.1083/jcb.142.6.1595. PMID 9744887
- ↑ Anderson DE, Losada A, Erickson HP, Hirano T. (2002). «Condensin and cohesin display different arm conformations with characteristic hinge angles». J. Cell Biol. 156 (6): 419—424. doi:10.1083/jcb.200111002. PMID 11815634
- ↑ Ono T, Fang Y, Spector DL, Hirano T (2004). "Spatial and temporal regulation of Condensins I and II in mitotic chromosome assembly in human cells". Mol. Biol. Cell 15 (7): 3296–308. doi:10.1091/mbc.E04-03-0242. PMID 15146063
- ↑ Hirota T, Gerlich D, Koch B, Ellenberg J, Peters JM (2004). "Distinct functions of condensin I and II in mitotic chromosome assembly". J. Cell Sci. 117 (Pt 26): 6435–45. doi:10.1242/jcs.01604. PMID 15572404
- ↑ Kimura K, Hirano M, Kobayashi R, Hirano T (1998). "Phosphorylation and activation of 13S condensin by Cdc2 in vitro". Science 282 (5388): 487–490. doi:10.1126/science.282.5388.487. PMID 9774278
- ↑ Johzuka K, Terasawa M, Ogawa H, Ogawa T, Horiuchi T (2006). "Condensin loaded onto the replication fork barrier site in the rRNA gene repeats during S phase in a FOB1-dependent fashion to prevent contraction of a long repetitive array in Saccharomyces cerevisiae.". Mol Cell Biol. 26 (6): 2226–2236. doi:10.1128/MCB.26.6.2226-2236.2006. PMID 16507999
- ↑ Haeusler RA, Pratt-Hyatt M, Good PD, Gipson TA, Engelke DR (2008). "Clustering of yeast tRNA genes is mediated by specific association of condensin with tRNA gene transcription complexes.". Genes Dev. 22 (16): 2204–2214. doi:10.1101/gad.1675908. PMID 18708579
- ↑ Hartl TA, Smith HF, Bosco G (2008). "Chromosome alignment and transvection are antagonized by condensin II.". Science 322 (5906): 1384–1387. doi:10.1126/science.1164216. PMID 19039137
- ↑ Bauer CR, Hartl TA, Bosco G. (2012). "Condensin II promotes the formation of chromosome territories by inducing axial compaction of polyploid interphase chromosomes.". PLoS Genet 8 (8): e1002873. doi:10.1371/journal.pgen.1002873. PMID 22956908
| ||||||||||||||||||||||||||||