Користувач:Julius sirius/Модель заміщення нуклеотидів
У біології модель заміщення нуклеотидів, яку також називають модель еволюції послідовності ДНК, — це модель Маркова, яка описує зміни протягом еволюційного часу. Ці моделі описують еволюційні зміни в макромолекулах (наприклад, послідовності ДНК ), представлених у вигляді послідовності символів (A, C, G і T у випадку ДНК ). Моделі заміщення використовуються для обчислення ймовірності філогенетичних дерев з використанням даних вирівнювання кількох послідовностей. Таким чином, моделі заміщення є центральними для оцінки максимальної правдоподібності філогенезу, а також баєсового висновування. Оцінки еволюційних відстаней (кількість замін, які відбулися після того, як пара послідовностей розійшлися від спільного предка) зазвичай розраховуються з використанням моделей заміщення або ж підстановки (еволюційні відстані використовуються як вхідні дані для методів розрахунку еволюційної відстані, таких як приєднання сусідів). Моделі підстановки також є центральними для філогенетичних інваріантів, оскільки їх можна використовувати для прогнозування частоти частот шаблону сайту з урахуванням топології дерева. Моделі заміни необхідні для моделювання даних послідовності для групи організмів, пов’язаних філогенетичними зв'язками.
Топології філогенетичного дерева та інші параметри[ред. | ред. код]
Топології філогенетичного дерева часто є параметром, який цікавить; [1] таким чином, довжина гілок та будь-які інші параметри, що описують процес заміни, часто розглядаються як некорисні параметри . Однак біологи іноді цікавляться іншими аспектами моделі. Наприклад, довжини гілок, особливо коли ці довжини гілок поєднуються з інформацією з літопису скам’янілостей і моделлю для оцінки часових рамок еволюції. [2] Інші параметри моделі були використані, щоб отримати уявлення про різні аспекти процесу еволюції. Відношення K <sub id="mwKg">a</sub> /K <sub id="mwKw">s</sub> (також зване ω в моделях заміщення кодонів) є параметром, який цікавить багатьох дослідників. Відношення K a /K s можна використовувати для дослідження дії природного добору на ділянки, що кодують білок; [3] він надає інформацію про відносні швидкості нуклеотидних замін, які змінюють амінокислоти (несинонімічні заміни) на ті, які не змінюють кодовану амінокислоту (синонімічні заміни).
Застосування до даних послідовності[ред. | ред. код]
Більша частина роботи над моделями заміни зосереджена на еволюції послідовності ДНК/ РНК та білка . Моделі еволюції послідовності ДНК, де алфавіт відповідає чотирьом нуклеотидам (A, C, G і T), є, мабуть, найпростішими моделями для розуміння. Моделі ДНК також можна використовувати для дослідження еволюції РНК-вірусу ; це відображає той факт, що РНК також має чотиринуклеотидний алфавіт (A, C, G і U). Проте моделі підстановки можна використовувати для алфавітів будь-якого розміру; алфавіт — це 20 протеїногенних амінокислот для білків і смислові кодони (тобто 61 кодон, який кодує амінокислоти в стандартному генетичному коді ) для вирівняних послідовностей генів, що кодують білок. Фактично, моделі заміщення можуть бути розроблені для будь-яких біологічних символів, які можуть бути закодовані за допомогою певного алфавіту (наприклад, амінокислотні послідовності в поєднанні з інформацією про конформацію цих амінокислот у тривимірних білкових структурах [4] ).
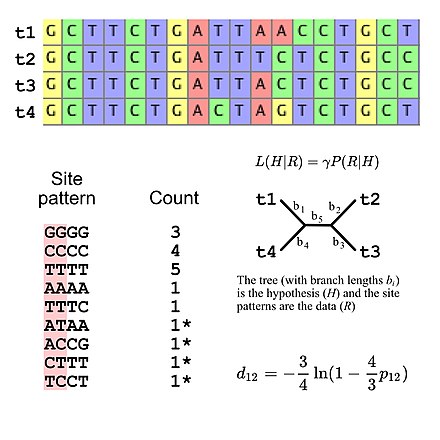
Більшість моделей заміщення, що використовуються для еволюційного дослідження, припускають незалежність між сайтами (тобто ймовірність спостереження будь-якого конкретного шаблону сайту ідентична незалежно від того, де знаходиться шаблон сайту у вирівнюванні послідовності). Це спрощує обчислення ймовірності, оскільки необхідно лише обчислити ймовірність усіх шаблонів сайту, які з’являються у вирівнюванні, а потім використати ці значення для обчислення загальної ймовірності вирівнювання (наприклад, ймовірність трьох шаблонів сайту «GGGG» за деякою моделлю еволюції послідовності ДНК – це просто ймовірність одного сайту «GGGG», піднята до третього ступеня). Це означає, що моделі підстановки можна розглядати як такі, що мають на увазі специфічний мультиноміальний розподіл для частот шаблонів сайту. Якщо ми розглянемо множинне вирівнювання послідовностей чотирьох послідовностей ДНК, то існує 256 можливих шаблонів сайтів, тож існує 255 ступенів свободи для частот шаблону сайту. Однак можна вказати очікувані частоти шаблону сайту, використовуючи п’ять ступенів свободи, якщо використовувати модель еволюції ДНК Джукса-Кантора [5], яка є простою моделлю заміни, яка дозволяє обчислювати очікувані частоти шаблону сайту для топології дерева та довжину гілок (враховуючи чотири таксони, неукорінене роздвоєне дерево має п’ять довжин гілок).
Моделі заміщення нуклеотидів також дозволяють моделювати дані послідовності за допомогою методів Монте-Карло . Змодельовані множинні вирівнювання послідовностей можна використовувати для оцінки ефективності філогенетичних методів [6] та створення нульового розподілу для певних статистичних тестів у галузях молекулярної еволюції та молекулярної філогенетики. Приклади цих тестів включають тести відповідності моделі [7] та «тест SOWH», який можна використовувати для дослідження топологій дерева. [8] [9]
Застосування до морфологічних даних[ред. | ред. код]
Той факт, що моделі заміщення можуть бути використані для аналізу будь-якого біологічного алфавіту, дає можливість розробити моделі еволюції для фенотипових наборів даних [10] (наприклад, морфологічних і поведінкових ознак). Як правило, "0". використовується для вказівки на відсутність ознаки, а «1» використовується для вказівки на наявність ознаки, хоча також можна оцінювати символи за допомогою кількох станів. Використовуючи цю структуру, ми можемо закодувати набір фенотипів як двійкові рядки (це можна узагальнити до рядків k -state для символів з більш ніж двома станами) перед аналізом за допомогою відповідного режиму. Це можна проілюструвати на прикладі «іграшки»: ми можемо використовувати двійковий алфавіт, щоб оцінити наступні фенотипові ознаки «має пір’я», «кладе яйця», «має хутро», «є теплокровним» і «здатний політ». У цьому прикладі іграшки колібрі матиме послідовність 11011 (більшість інших птахів матиме таку саму нитку), страуси – 11010, велика рогата худоба (та більшість інших наземних ссавців ) – 00110, а кажани – 00111. Імовірність філогенетичного дерева потім може бути розрахована за допомогою цих бінарних послідовностей і відповідної моделі заміни. Існування цих морфологічних моделей дає змогу аналізувати матриці даних з викопними таксонами, використовуючи лише морфологічні дані [11] або комбінацію морфологічних і молекулярних даних [12] (при цьому останні були оцінені як відсутні дані для викопних таксонів). .
Існує очевидна схожість між використанням молекулярних або фенотипічних даних у сфері кладистики та аналізом морфологічних ознак за допомогою моделі заміщення. Однак у суспільстві систематичних систем точилися гучні дебати [a] щодо питання про те, чи слід розглядати кладистичний аналіз як «безмодельний». Сфера кладистики (визначена в найсуворішому сенсі) сприяє використанню критерію максимальної парсимонії для філогенетичного висновку. [13] Багато кладистів відкидають позицію про те, що максимальна парсимонія заснована на моделі заміни, і (у багатьох випадках) вони виправдовують використання економії, використовуючи філософію Карла Поппера . [14] Однак існування моделей «еквівалентної парсимонії» [15] (тобто моделей підстановки, які дають максимальне дерево парсимонії при використанні для аналізу) дає змогу розглядати парсимонію як модель підстановки. [1]
Молекулярний годинник і одиниці часу[ред. | ред. код]
Як правило, довжина гілки філогенетичного дерева виражається як очікувана кількість замін на сайт; якщо еволюційна модель вказує, що кожен сайт у родовій послідовності зазвичай зазнає x замін до того часу, коли він еволюціонує до послідовності конкретного нащадка, то предок і нащадок вважаються розділеними довжиною гілки x .
Іноді довжину гілки вимірюють у геологічних роках. Наприклад, дані скам’янілостей можуть дати можливість визначити кількість років між видом предків і видами-нащадками. Оскільки деякі види еволюціонують швидше, ніж інші, ці два показники довжини гілок не завжди знаходяться в прямій пропорції. Очікувана кількість замін на сайт на рік часто позначається грецькою літерою mu (μ).
Вважається, що модель має суворий молекулярний годинник, якщо очікувана кількість замін на рік μ є постійною незалежно від того, еволюція якого виду досліджується. Важливим наслідком суворого молекулярного годинника є те, що кількість очікуваних замін між родовим видом і будь-яким із його сучасних нащадків має бути незалежною від того, який вид нащадків досліджується.
Зауважте, що припущення про суворий молекулярний годинник часто нереалістичне, особливо протягом тривалих періодів еволюції. Наприклад, незважаючи на те, що гризуни генетично дуже схожі на приматів, вони зазнали набагато більшої кількості замін за оцінений час після розбіжності в деяких областях геному. [16] Це може бути пов’язано з меншим часом генерації, [17] більш високою швидкістю метаболізму, збільшеною структурою популяції, збільшенням швидкості видоутворення або меншим розміром тіла . [18] [19] При вивченні давніх подій, таких як кембрійський вибух, за припущенням молекулярного годинника, часто спостерігається низький збіг між кладистичними та філогенетичними даними. Існує кілька досліджень щодо варіабельності швидкість еволюції. [20] [21]
Моделі, які можуть враховувати мінливість швидкості молекулярного годинника між різними еволюційними лініями у філогенезі, називаються «розслабленими» на противагу «суворим». У таких моделях можна вважати, що швидкість корелює чи ні між предками та нащадками, а варіація швидкості між родовідами може бути отримана з багатьох розподілів, але зазвичай застосовуються експоненційні та логнормальні розподіли. Існує окремий випадок, який називається «локальним молекулярним годинником», коли філогенез поділено щонайменше на два розділи (набори родовищ) і в кожному використовується строгий молекулярний годинник, але з різними темпами.
Реверсивні відносно часу та стаціонарні моделі[ред. | ред. код]
Багато корисних моделей заміни є оборотними в часі; з точки зору математики, для моделі не має значення, яка послідовність є предком, а яка нащадком, доки всі інші параметри (наприклад, кількість замін на сайт, яка очікується між двома послідовностями) залишаються незмінними.
При аналізі реальних біологічних даних, як правило, немає доступу до послідовностей предків, а лише до сучасних видів. Однак, коли модель є оборотною в часі, який вид був родовим, не має значення. Натомість філогенетичне дерево можна вкорінити за допомогою будь-якого з видів, пізніше вкорінити на основі нових знань або залишити без коренів. Це тому, що не існує «особливих» видів, усі види в кінцевому підсумку походять один від одного з однаковою ймовірністю.
Модель є оборотною в часі тоді і тільки тоді, коли вона задовольняє властивість (позначення пояснюється нижче)
або, еквівалентно, властивість детального балансу ,
для кожного i, j і t .
Зворотність у часі не слід плутати зі стаціонарністю. Модель є нерухомою, якщо Q не змінюється з часом. Аналіз нижче передбачає стаціонарну модель.
Математика моделей заміщення нуклеотидів[ред. | ред. код]
[[Категорія:Стохастичні моделі]] [[Категорія:Біоінформатика]] [[Категорія:Сторінки із неперевіреними перекладами]]
- ↑ а б Steel M, Penny D (June 2000). Parsimony, likelihood, and the role of models in molecular phylogenetics. Molecular Biology and Evolution. 17 (6): 839—50. doi:10.1093/oxfordjournals.molbev.a026364. PMID 10833190. Помилка цитування: Некоректний тег
<ref>; назва «:4» визначена кілька разів з різним вмістом - ↑ Bromham L (May 2019). Six Impossible Things before Breakfast: Assumptions, Models, and Belief in Molecular Dating. Trends in Ecology & Evolution. 34 (5): 474—486. doi:10.1016/j.tree.2019.01.017. PMID 30904189.
- ↑ Yang Z, Bielawski JP (December 2000). Statistical methods for detecting molecular adaptation. Trends in Ecology & Evolution. 15 (12): 496—503. doi:10.1016/s0169-5347(00)01994-7. PMC 7134603. PMID 11114436.
- ↑ Perron U, Kozlov AM, Stamatakis A, Goldman N, Moal IH (September 2019). Modeling Structural Constraints on Protein Evolution via Side-Chain Conformational States. Molecular Biology and Evolution. 36 (9): 2086—2103. doi:10.1093/molbev/msz122. PMC 6736381. PMID 31114882.
- ↑ Evolution of Protein Molecules. Mammalian Protein Metabolism. Т. 3. Elsevier. 1969. с. 21—132. doi:10.1016/b978-1-4832-3211-9.50009-7. ISBN 978-1-4832-3211-9.
- ↑ Huelsenbeck JP, Hillis DM (1 вересня 1993). Success of Phylogenetic Methods in the Four-Taxon Case. Systematic Biology (англ.). 42 (3): 247—264. doi:10.1093/sysbio/42.3.247. ISSN 1063-5157.
- ↑ Goldman N (February 1993). Statistical tests of models of DNA substitution. Journal of Molecular Evolution. 36 (2): 182—98. Bibcode:1993JMolE..36..182G. doi:10.1007/BF00166252. PMID 7679448.
- ↑ Swofford D.L. Olsen G.J. Waddell P.J. Hillis D.M. 1996. "Phylogenetic inference." in Molecular systematics (ed. Hillis D.M. Moritz C. Mable B.K.) 2nd ed. Sunderland, MA: Sinauer. p. 407–514. ISBN 978-0878932825
- ↑ Church SH, Ryan JF, Dunn CW (November 2015). Automation and Evaluation of the SOWH Test with SOWHAT. Systematic Biology. 64 (6): 1048—58. doi:10.1093/sysbio/syv055. PMC 4604836. PMID 26231182.
- ↑ Lewis PO (1 листопада 2001). A likelihood approach to estimating phylogeny from discrete morphological character data. Systematic Biology. 50 (6): 913—25. doi:10.1080/106351501753462876. PMID 12116640.
- ↑ Lee MS, Cau A, Naish D, Dyke GJ (May 2014). Morphological clocks in paleontology, and a mid-Cretaceous origin of crown Aves. Systematic Biology. 63 (3): 442—9. doi:10.1093/sysbio/syt110. PMID 24449041.
- ↑ Ronquist F, Klopfstein S, Vilhelmsen L, Schulmeister S, Murray DL, Rasnitsyn AP (December 2012). A total-evidence approach to dating with fossils, applied to the early radiation of the hymenoptera. Systematic Biology. 61 (6): 973—99. doi:10.1093/sysbio/sys058. PMC 3478566. PMID 22723471.
- ↑ Brower, A. V .Z. (2016). "Are we all cladists?" in Williams, D., Schmitt, M., & Wheeler, Q. (Eds.). The future of phylogenetic systematics: The legacy of Willi Hennig (Systematics Association Special Volume Series Book 86). Cambridge University Press. pp. 88-114 ISBN 978-1107117648
- ↑ Farris JS, Kluge AG, Carpenter JM (1 травня 2001). Olmstead R (ред.). Popper and Likelihood Versus "Popper*". Systematic Biology (англ.). 50 (3): 438—444. doi:10.1080/10635150119150. ISSN 1076-836X. PMID 12116585.
- ↑ Goldman, Nick (December 1990). Maximum Likelihood Inference of Phylogenetic Trees, with Special Reference to a Poisson Process Model of DNA Substitution and to Parsimony Analyses. Systematic Zoology. 39 (4): 345—361. doi:10.2307/2992355. JSTOR 2992355.
- ↑ Gu X, Li WH (September 1992). Higher rates of amino acid substitution in rodents than in humans. Molecular Phylogenetics and Evolution. 1 (3): 211—4. doi:10.1016/1055-7903(92)90017-B. PMID 1342937.
- ↑ Li WH, Ellsworth DL, Krushkal J, Chang BH, Hewett-Emmett D (February 1996). Rates of nucleotide substitution in primates and rodents and the generation-time effect hypothesis. Molecular Phylogenetics and Evolution. 5 (1): 182—7. doi:10.1006/mpev.1996.0012. PMID 8673286.
- ↑ Martin AP, Palumbi SR (May 1993). Body size, metabolic rate, generation time, and the molecular clock. Proceedings of the National Academy of Sciences of the United States of America. 90 (9): 4087—91. Bibcode:1993PNAS...90.4087M. doi:10.1073/pnas.90.9.4087. PMC 46451. PMID 8483925.
- ↑ Yang Z, Nielsen R (April 1998). Synonymous and nonsynonymous rate variation in nuclear genes of mammals. Journal of Molecular Evolution. 46 (4): 409—18. Bibcode:1998JMolE..46..409Y. CiteSeerX 10.1.1.19.7744. doi:10.1007/PL00006320. PMID 9541535.
- ↑ Kishino H, Thorne JL, Bruno WJ (March 2001). Performance of a divergence time estimation method under a probabilistic model of rate evolution. Molecular Biology and Evolution. 18 (3): 352—61. doi:10.1093/oxfordjournals.molbev.a003811. PMID 11230536.
- ↑ Thorne JL, Kishino H, Painter IS (December 1998). Estimating the rate of evolution of the rate of molecular evolution. Molecular Biology and Evolution. 15 (12): 1647—57. doi:10.1093/oxfordjournals.molbev.a025892. PMID 9866200.
Помилка цитування: Теги <ref> існують для групи під назвою «lower-alpha», але не знайдено відповідного тегу <references group="lower-alpha"/>

