Повне секвенування геному


Повне секвенування генома (англ. Whole genome sequencing, або WGS) — це процес одноразового визначення всієї або майже всієї послідовності ДНК геному організму.[2] Він передбачає секвенування всієї хромосомної ДНК організму, а також ДНК, що міститься в мітохондріях, а для рослин також у хлоропласті.
Повне секвенування генома в основному використовувалося як інструмент дослідження в наукових цілях, але також було консультативно введено в лікарні США для виявлення причин захворювань та методів їх лікування з 2014 року.[3][4][5] У майбутньому при широкому застосуванні персоналізованої медицини дані про послідовність повного геному можуть бути важливим інструментом для терапевтичного втручання.[6] Секвенування генів на рівні однонуклеотидного поліморфізму також використовується для точного визначення функціональних певних варіантів із генетичних асоціацій та покращення знань, доступних дослідникам, які цікавляться еволюційною біологією, і, отже, може закласти основу для прогнозування сприйнятливості до захворювань і реакції на ліки.
Повне секвенування генома не слід плутати з генетичним фінґерпринтингом, яке лише визначає ймовірність того, що генетичний матеріал походить від конкретної особи чи групи, і не містить додаткової інформації про генетичні зв'язки, походження чи схильність до конкретних захворювань.[7] Крім того, повне секвенування геному не слід плутати з методами, які секвенують конкретні підмножини геному — такі методи включають секвенування всього екзома (1–2 % геному) або генотипування.





Методи секвенування ДНК, які використовувалися в 1970-х і 1980-х роках, були ручними; наприклад, такі методи як секвенування Максама–Гілберта та секвенування Сенгера. Кілька цілих геномів бактеріофагів і вірусів тварин були секвеновані за допомогою цих методів, але перехід до більш швидких, автоматизованих методів секвенування в 1990-х роках полегшив секвенування більших бактеріальних і еукаріотичних геномів.[9]
Першим вірусом, з повністю секвенованим геном, був бактеріофаг MS2 у 1976 році.[10] У 1992 році хромосома III була першою хромосомою будь-якого організму, яка була повністю секвенована.[11] Першим організмом, чий геном був повністю секвенований, був Haemophilus influenzae у 1995 році.[12] Після цього вперше були секвеновані геноми інших бактерій і деяких архей, головним чином через їх малий розмір геному. Наприклад, H. influenzae має геном лише з 1 830 140 пар основ ДНК.[12] Навпаки, еукаріоти, як одноклітинні, так і багатоклітинні, такі як Amoeba dubia та люди (Homo sapiens) відповідно, мають набагато більші геноми (див. Парадокс C-значення).[13] Amoeba dubia має геном із 700 мільярдів пар нуклеотидів, розподілених по тисячах хромосом.[14] Людина ж містить менше пар нуклеотидів (приблизно 3,2 мільярда в кожній клітині), однак розмір їх геному значно перевищує розмір геному окремих бактерій.[15]
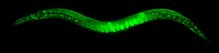
Перші бактеріальні та архейні геноми, включно з геномом H. influenzae, були секвеновані методом секвенування Shotgun .[12] У 1996 році було секвеновано перший еукаріотичний геном пивних дріждів (Saccharomyces cerevisiae). Також, S. cerevisiae, який визначено як модельний організм у біології, має геном лише з 12 мільйонів пар нуклеотидів,[16] , був першим одноклітинним еукаріотом, у якого повністю секвенували геном. Першим багатоклітинним еукаріотом і твариною, чий геном був секвенований, був нематодний черв'як: Caenorhabditis elegans у 1998 році.[17] Еукаріотичні геноми секвенуються декількома методами, включаючи секвенування коротких фрагментів ДНК за допомогою дробовика та секвенування більших клонів ДНК з бібліотек ДНК, таких як бактеріальні штучні хромосоми (BAC) і дріжджових штучних хромосом (YAC).[18]
У 1999 році була опублікована повна послідовність ДНК 22-ї хромосоми людини, другої найкоротшої аутосоми людини.[19] До 2000 року було секвеновано геном другої тварини та другого безхребетного (але першої комахи) — плодової мушки Drosophila melanogaster — популярного вибору модельного організму в експериментальних дослідженнях.[20] Перший геном рослини — модельного організму Arabidopsis thaliana — також був повністю секвенований до 2000 року.[21] Геном лабораторної миші Mus musculus був завершений у 2002 році[22] .
У 2004 році Human Genome Project опублікував неповну версію геному людини.[23] У 2008 році група з Лейдена, Нідерланди, повідомила про секвенування геному першої жінки (Марджолейн Крік).
На даний момент тисячі геномів повністю або частково секвеновані.
Майже будь-який біологічний зразок, що містить повну копію ДНК, або дуже невелику кількість ДНК або стародавньої ДНК — може надати генетичний матеріал, необхідний для секвенування геному. Зразками можуть бути: слина, епітеліальні клітини, кістковий мозок, волосся (за умови, що волосся містить волосяний фолікул), насіння, листя рослин або будь-що інше, що містить ті клітини, які містять ДНК.
Геном однієї клітини, вибраної із популяції генетично різних клітин, може бути визначена за допомогою методів секвенування геному однієї клітини. Це має застосування в мікробіології у випадках, коли одну клітину певного виду мікроорганізму можна виділити із популяції за допомогою мікроскопії на основі її морфологічних або інших відмінних характеристик. У таких випадках зазвичай необхідні етапи ізоляції та вирощування організму в культурі можуть бути опущені, що дозволяє секвенувати набагато більший спектр геномів організму.[24]
Секвенування генома однієї клітини випробовується як метод передімплантаційної генетичної діагностики, коли клітина ембріона, створеного шляхом штучного запліднення, береться та аналізується перед перенесенням ембріона в матку.[25] Після імплантації безклітинну фетальну ДНК можна взяти простою венепункцією у матері та використати для секвенування повного генома плоду.[26]

Секвенування майже всього геному людини було вперше виконано в 2000 році частково за допомогою технології секвенування дробовика. У той час як повне геномне секвенування для невеликих (4000–7000 пар основ) геномів уже використовувалося в 1979 році,[27] більш широке застосування виграло від попарного кінцевого секвенування, відомого в розмовній мові як двоствольне секвенування . Коли проекти секвенування почали охоплювати довші та складніші геноми, багато груп почали усвідомлювати, що корисну інформацію можна отримати шляхом секвенування обох кінців фрагмента ДНК. Хоча секвенування обох кінців одного фрагмента та відстеження парних даних було більш громіздким, ніж секвенування одного кінця двох різних фрагментів, знання того, що дві послідовності були орієнтовані в протилежних напрямках і були приблизно довжиною фрагмента окремо від кожного інше було цінним у реконструкції послідовності оригінального цільового фрагмента.
Перший опублікований опис використання ланцюжків був опублікований в 1990 році в рамках секвенування локусу HPRT у людини[28]. Перший теоретичний опис стратегії чистого попарного секвенування ланцюжків, що передбачає використання фрагментів постійної довжини, було опубліковано в 1991 році[29].У 1995 році було введено нововведення у вигляді використання фрагментів різного розміру[30], яке продемонструвало, що стратегія чистого попарного секвенування ланцюжків може бути реалізована на великих об'єктах. Згодом ця стратегія була прийнята Інститутом геномних досліджень (англ. The Institute for Genomic Research[en] TIGR) для секвенування всього геному бактерії Haemophilus influenzae у 1995 році[31], а потім компанією Celera Genomics для секвенування всього геному плодової мушки в 2000 році[32], а потім і всього геному людини. Компанія Applied Biosystems, яка тепер називається Life Technologies, виготовила автоматизовані Капілярні секвенсори, що використовуються як Celera Genomics, так і проектом «Геном людини».
Хоча капілярне секвенування було першим підходом до успішного секвенування майже повного геному людини, воно до цього часу все ще занадто дороге та займає надто багато часу для цілей широкого застосування. З 2005 року капілярне секвенування поступово витісняється Масово-паралельним секвенуванням (раніше відомим під назвою «секвенування наступного покоління» «next-generation sequencing») та новітніми технологіями секвенування, такими як Метод Illumina/Solexa, піросеквенування та секвенування SMRT.[33] Усі ці технології продовжують використовувати базову стратегію секвенування «дробовика», а саме розпаралелювання процесу та створення шаблонів розпізнавання за допомогою фрагментації геному.
З'явилися також інші технології, зокрема технологія нанопор. Хоча точність секвенування технології нанопор нижча, ніж у наведених вище технологіях, її довжина зчитування в середньому набагато довша.[34] Ця генерація довгих зчитувань є цінною особливістю технології, особливо в програмах секвенування всього генома заново.[35]
Повне секвенування генома (анг. Whole genome sequencing, або WGS) — це процес одноразового визначення всієї або майже всієї послідовності ДНК геному організму.[36] Він передбачає секвенування всієї хромосомної ДНК організму, а також ДНК, що міститься в мітохондріях, а для для рослин також у хлоропласті.
Оскільки процес секвенування генерує багато даних (наприклад, у кожному диплоїдному геномі людини є приблизно шість мільярдів пар основ), його результати зберігаються в електронному вигляді та потребують великої обчислювальної потужності для аналізу та пам'яті для зберігання.
Хоча аналіз даних повного геномного секвенування може бути повільним, цей крок можна пришвидшити за допомогою спеціального обладнання.[37]

Низка державних і приватних компаній змагаються за розробку платформи секвенування геному, яка є комерційно надійною як для досліджень, так і для клінічного використання,[38] включаючи такі компанії як Illumina,[39] Knome,[40] Sequenom,[41] 454 Life Sciences,[42] Pacific Biosciences,[43] Complete Genomics,[44] Helicos Biosciences,[45] GE Global Research (General Electric), Affymetrix, IBM, Intelligent Bio-Systems,[46] Life Technologies, Oxford Nanopore Technologies,[47] та Пекінський інститут геноміки.[48][49][50] Ці компанії та установи значною мірою фінансуються та підтримуються венчурним капіталом, хедж-фондами та інвестиційними банками.[51][52]
До кінця 2010-х років комерційна цільова вартість секвенування становила 1000 доларів США. США, однак приватні компанії працюють над досягненням нової цілі лише в 100 дол.[53]
У жовтні 2006 року Фонд X Prize, який співпрацює з Науковим фондом Дж. Крейга Вентера, заснував Archon X Prize for Genomics[54] маючи намір присудити 10 мільйонів доларів «першій команді, яка зможе створити пристрій і використати його для секвенування 100 геномів людини протягом 10 днів або менше з точністю не більше однієї помилки на кожні 1 000 000 секвенованих основ, причому послідовності точно охоплюють щонайменше 98 % геному, і з постійною вартістю не більше 1000 доларів США за геном».[55] Премія Archon X У жовтні 2006 року Фонд X Prize, який співпрацює з Науковим фондом Дж. Крейга Вентера, заснував Archon X Prize for Genomics[56] маючи намір присудити 10 мільйонів доларів «першій команді, яка зможе створити пристрій і використати його для секвенування 100 геномів людини протягом 10 днів або менше з точністю не більше однієї помилки на кожні 1 000 000 секвенованих основ, причому послідовності точно охоплюють щонайменше 98 % геному, і з постійною вартістю не більше 1000 доларів США за геном».[57] Prize з геноміки була скасована в 2013 році, до офіційної дати її початку.[58][59]
У 2007 році Applied Biosystems почала продавати новий тип секвенсора під назвою SOLiD System.[60] Технологія дозволяла користувачам секвенувати 60 гігаоснов за один запуск.[61]
У червні 2009 року компанія Illumina оголосила про запуск власної програми Personal Full Genome Sequencing Service на глибину 30× за 48 000 доларів США за геном.[62][63] У серпні засновник Helicos Biosciences Стівен Куейк заявив, що за допомогою Single Molecule Sequencer своєї компанії він секвенував власний геном повністю менш ніж за 50 000 доларів.[64] У листопаді Complete Genomics опублікувала рецензовану статтю в Science, демонструючи свою здатність секвенувати повний геном людини за 1700 доларів.[65][66]
У травні 2011 року Illumina знизила вартість послуги повного секвенування геному до 5000 доларів США за геном людини або 4000 доларів США за замовлення 50 і більше проектів.[67] Helicos Biosciences, Pacific Biosciences, Complete Genomics, Illumina, Sequenom, ION Torrent Systems, Halcyon Molecular, NABsys, IBM і GE Global, схоже, ведуть боротьбу за комерціалізацію повного секвенування геному.[33][68]
Зі зниженням витрат на секвенування ряд компаній почали стверджувати, що їхнє обладнання незабаром досягне генома в 1000 доларів США: У 2015 році NHGRI оцінив вартість отримання послідовності всього генома людини приблизно в 1500 доларів.[69] У 2016 році Veritas Genetics почала продавати таку послугу як повне секвенування геному для широкого загалу, включаючи звіт про частину інформації в секвенуванні за 999 доларів.[70] Влітку 2019 року Veritas Genetics знизила вартість послуги до 599 доларів.[71] У 2017 році BGI почала пропонувати таку послугу як повне секвенування геному для широкого загалу за 600 доларів.[72]
Однак у 2015 році деякі аналітики зауважили, що ефективне визначення та аналіз повністю секвенованого генома може коштувати значно більше ніж 1000 доларів.[73] Крім того, як повідомляється, залишаються частини геному людини, які не були повністю секвеновані до 2017 року.[74][75]
Технологія повного секвенування генома надає інформацію про геном в об'ємі пам'яті на порядки більшу, ніж попередня технологія використання мікрочипів ДНК.
Для людей, технологія мікрочипів ДНК наразі надає генотипову інформацію до одного мільйона генетичних варіантів[76][77][78], тоді як технологія повного секвенування геному надає інформацію про всі шість мільярдів основ у геномі людини, або це в 3000 разів більше даних. Через це повне секвенування геному вважається революційним нововведенням на ринку мікрочипів ДНК, оскільки точність обох коливається від 99,98 % до 99,999 % (у неповторюваних областях ДНК), а вартість витратних матеріалів у 5000 доларів США за 6 мільярдів пар основ є конкурентоспроможною. в порівнянні з мікрочипами ДНК (500 доларів США за 1 мільйон пар основ).[42]
Повне секвенування геному дало змогу встановити частоту мутацій для цілих геномів людини. Частота мутацій у всьому геномі між поколіннями людини (від батьків до дитини) становить близько 70 нових мутацій на покоління.[79][80] Ще нижчий рівень варіації (тобто, мутацій) було виявлено при порівняльному секвенуванні геному з клітинах крові пари монозиготних (ідентичних близнюків) 100-річних довгожителів.[81] Було виявлено лише 8 соматичних відмінностей, хоча соматичні варіації, що відбуваються в менш ніж 20 % клітин крові, не могли бути виявлені.
За деякими оцінками, у ділянках генома людини, що кодують білок, існує приблизно 0,35 мутації, які можуть змінити послідовність білка між поколіннями батьків/нащадків (менше одного мутованого білка на покоління).[82]
В ракових клітинах частота мутацій набагато вища через нестабільність геному. Ця частота також може залежати від віку пацієнта, впливу агентів, що пошкоджують ДНК (таких як УФ-опромінення або компоненти тютюнового диму), і активності/неактивності механізмів відновлення ДНК. Крім того, частота мутацій може відрізнятися для ракових клітин: у клітинах зародкової лінії частота мутацій становить приблизно 0,023 мутації на мегаоснови, але ця кількість набагато вища при раку молочної залози (1,18-1,66 соматичних мутацій на Mb), при раку легенів (17,7) або при меланомах (≈33).[83] Оскільки гаплоїдний геном людини складається приблизно з 3200 мегаоснов[84], це означає приблизно 74 мутації (переважно в некодуючих ділянках) у зародковій ДНК за покоління, але 3776-5312 соматичних мутацій на гаплоїдний геном при раку молочної залози, 56 640 при раку легенів і 105 600 у меланомах.
Розподіл соматичних мутацій у геномі людини дуже нерівномірний[85], так що багаті на гени ділянки ранньої реплікації отримують менше мутацій, ніж гетерохроматин із бідним геном і пізньою реплікацією, ймовірно, через диференціальну активність відновлення ДНК.[86] Зокрема, модифікація гістону H3K9me3 асоціюється з високою,[87] а H3K36me3 з низькою частотою мутацій.[88]
У дослідженнях повногеномне секвенування можна використовувати в дослідженні загальногеномної асоціації (англ. Genome-Wide Association Study, GWAS) — проекті, спрямованому на визначення генетичного варіанту або варіантів, пов'язаних із захворюванням або іншим фенотипом.[89]
У 2009 році Illumina випустила свої перші секвенатори повного генома, які були випробувані для клінічного використання, а не лише для використання в дослідженнях, і лікарі в академічних медичних центрах почали використовувати їх, щоб спробувати діагностувати, що не так з людьми, яким стандартні підходи не допомагали.[90] У 2009 році команда зі Стенфорда під керівництвом Юана Ешлі виконала клінічну інтерпретацію повного геному людини, біоінженера Стівена Куейка.[91] У 2010 році команда Ешлі повідомила про молекулярний розтин цілого генома[92], а в 2011 році розширила структуру інтерпретації до повністю секвенованої сім'ї, сім'ї Вест, яка була першою сім'єю, яку секвенували на платформі Illumina.[93] Ціна секвенування геному на той час становила 19 500 доларів доларів США, які виставлялися пацієнту як оплата за медичні послуги, але зазвичай оплачувалися за рахунок гранту на дослідження. Варто відзначити, що одна особа в той час подала заявку на відшкодування від своєї страхової компанії.[90]
Наприклад, одній дитині знадобилося близько 100 операцій, коли їй виповнилося три роки, і її лікар звернувся до секвенування всього геному, щоб визначити проблему; для виявлення рідкісної мутації в XIAP, яка спричиняла масові проблеми, знадобилася команда з приблизно 30 осіб, до складу якої входили 12 експертів з біоінформатики, три спеціалісти з секвенування, п'ять лікарів, два генетичних консультанта та два фахівці з етики.[90][94][95]
Завдяки нещодавньому зниженню вартості повного секвенування генома застосування цього методу діагностики ДНК в лікуванні стало реалістичним. У 2013 році консорціум 3Gb-TEST отримав фінансування від Європейського Союзу для підготовки системи охорони здоров'я до цих інновацій у ДНК-діагностиці.[96][97] Згідно з вимогами, мають існувати такі настанови для впровадження: оцінка якості, оцінка технологій охорони здоров'я та керівні принципи. Консорціум 3Gb-TEST визначив аналіз та інтерпретацію даних після секвенування як найскладніший крок у діагностичному процесі.[98] На зустрічі Консорціуму в Афінах у вересні 2014 року Консорціум винайшов слово генотрансляція для цього важливого кроку. Цей крок веде до так званого генорепорту. А щоб визначити примінення таких звітів генорепорту, необхідні рекомендації.
Була також організована Genomes2People (G2P), ініціатива Brigham and Women's Hospital і Гарвардської медичної школи. Вона створена в 2011 році для вивчення інтеграції геномного секвенування в клінічну допомогу дорослим і дітям.[99]
У 2018 році дослідники з Інституту геномної медицини дитячої лікарні Рейді в Сан-Дієго визначили, що швидке повне секвенування геному (англ. rapid whole-genome sequencing, rWGS) може вчасно діагностувати генетичні розлади, щоб замінити гостру медичну або хірургічну терапію і покращити результати у гострохворих немовлят. У ретроспективному дослідженні гострохворих немовлят в обласній дитячій лікарні з липня 2016 року по березень 2017 року сорок дві сім'ї отримали rWGS для етіологічної діагностики генетичних розладів. Діагностична чутливість rWGS становила 43 % (вісімнадцять із 42 немовлят) і 10 % (четверо з 42 немовлят) для стандартних генетичних тестів (P = 0,0005). Показник клінічної користі rWGS (31 %, тринадцять із 42 немовлят) був значно вищим, ніж для стандартних генетичних тестів (2 %, один із 42; P = 0,0015). Одинадцять (26 %) немовлят з діагностичним rWGS уникли захворювання, у одного було зниження ймовірності смерті на 43 %, а одне розпочало паліативну допомогу. У шести з одинадцяти немовлят зміни в керівництві по догляду зменшили вартість стаціонарного лікування на 800 000-2 000 000 доларів США. Результати повторили попереднє дослідження клінічної користі rWGS у гострохворих стаціонарних немовлят і продемонстрували покращення результатів, чисту економію на охороні здоров'я.[100]
У 2018 році огляд 36 публікацій показав, що вартість повного секвенування генома коливається від 1906 доларів США. доларів США до 24 810 доларів США США і має широкий діапазон у діагностичній ефективності від 17 % до 73 % залежно від групи пацієнтів.[101]
Дослідження повного секвенування генома дають змогу оцінити зв'язки між складними ознаками та кодуючими та некодуючими рідкісними варіантами (частота малої алелі (англ. minor allele frequency[en], MAF) < 1 %) у всьому геномі. Одноваріантний аналіз (тобто аналіз, спрямований лише на 1 можливий варіант) зазвичай має низьку результативність для виявлення зв'язків з іншими рідкісними варіантами для визначення всієї множини варіантів, які кодують саме це захворювання. Тому були запропоновані тести набору варіантів для спільного тестування кумулятивних ефектів кількох рідкісних варіантів.[102][103][104]
Метааналіз повного секвенування генома виглядає привабливо для вирішення проблеми збору великих розмірів вибірки з метою виявлення рідкісних варіантів, пов'язаних зі складними фенотипами.
Повне секвенування геному надає великий набір викликів, з якими стикається наукове співтовариство, для аналізу, кількісного визначення та охарактеризування циркулюючої пухлинни ДНК (ctDNA) у крові. Це служить основою для ранньої діагностики раку, вибору лікування та моніторингу рецидивів, а також для визначення механізмів резистентності, метастазування та філогенетичних моделей в еволюції раку. Це також може допомогти у виборі індивідуальних методів лікування для пацієнтів, які страждають на цю патологію, і спостерігати за тим, як існуючі препарати діють під час прогресування лікування.
У 2013 році Грін і команда дослідників запустили проект BabySeq для вивчення етичних і медичних наслідків секвенування ДНК новонароджених.[105][106] Станом на 2015 рік обговорювалося повногеномне та екзомне секвенування як інструмент скринінгу новонароджених[107], і у 2021 році воно було додатково обговорене.[108]
У 2021 році фонд NIH профінансував BabySeq2, дослідження, яке розширило проект BabySeq, залучивши 500 немовлят з різних сімей і відстежуючи вплив їхнього геномного секвенування на педіатричну допомогу.[109]
У 2023 році журнал the Lancet висловив думку, що у Великобританії «фокусування на покращенні скринінгу шляхом оновлення цільових генів може бути більш розумним у короткостроковій перспективі. Секвенування всього генома в довгостроковій перспективі заслуговує ретельного вивчення та загальної обережності».[110]
Запровадження повного секвенування геному може мати етичні наслідки.[111] З одного боку, генетичне тестування може потенційно діагностувати хвороби, яким можна запобігти, як у особи, яка проходить генетичне тестування, так і в її родичів.[111] З іншого боку, генетичне тестування має потенційні недоліки, такі як генетична дискримінація, втрата анонімності та психологічний вплив, наприклад виявлення відсутності батьківства у чоловіків, які виховують дітей.[112]
Деякі поборники етики наполягають на тому, що конфіденційність осіб, які проходять генетичне тестування, має бути захищена[111], і це викликає особливе занепокоєння, коли генетичне тестування проходять неповнолітні.[113] Генеральний директор Illumina, Джей Флетлі, заявив у лютому 2009 року, що «до 2019 року редагування генів немовлят під час їх народження стане рутиною».[114] Це потенційне використання механізму секвенування генома є дуже суперечливим, оскільки воно протирічить встановленим етичним нормам прогностичного генетичного тестування безсимптомних неповнолітніх, які добре закріплені в галузі медичної генетики та генетичного консультування.[115][116][117][118]
Коли людина проходить повне секвенування геному, вона розкриває інформацію не лише про свої власні послідовності ДНК, але й про ймовірні послідовності ДНК своїх близьких генетичних родичів.[111] Ця інформація може додатково розкрити корисну прогнозну інформацію про теперішні та майбутні ризики для здоров'я родичів.[119] Отже, виникають важливі питання про те, які права, якщо такі є, мають члени сім'ї осіб, які генетично тестуються. У західному/європейському суспільстві тестованих осіб зазвичай заохочують ділитися важливою інформацією про будь-які генетичні діагнози зі своїми близькими родичами, оскільки важливість генетичного діагнозу для нащадків та інших близьких родичів зазвичай є однією з причин звернення для генетичного тестуванням у першу чергу.[111] Тим не менш, може розвинутися серйозна етична дилема, коли пацієнти відмовляються ділитися інформацією про діагноз, який свідчить про серйозний генетичний розлад і якому можна запобігти, та про існування високого ризику для родичів щодо цього діагнозу і які є носіями чи можуть бути носіями такої ж мутації . За таких обставин клініцист може підозрювати, що родичі воліють знати про діагноз, і, отже, клініцист може зіткнутися з конфліктом інтересів щодо конфіденційності між пацієнтом і лікарем.[111]
Занепокоєння щодо конфіденційності також може виникнути, коли повне секвенування геному використовується в наукових дослідженнях. Дослідникам часто доводиться поміщати інформацію про генотипи та фенотипи пацієнтів у загальнодоступні наукові бази даних, такі як бази даних щодо локусів.[111] Незважаючи на те, що лише анонімні дані про пацієнтів надсилаються до баз даних для специфічних локусів, родичі все одно можуть ідентифікувати пацієнтів у разі виявлення рідкісної хвороби або рідкісної місенс-мутації.[111] Також, публічна дискусія щодо запровадження передових криміналістичних методів (таких як просунутий пошук сім'ї з використанням загальнодоступних веб-сайтів походження ДНК та підходи до фенотипування ДНК) була обмеженою, розрізненою та нецілеспрямованою. У міру того, як судово-медична генетика зближується до секвенування геному, проблеми, пов'язані з генетичними даними, стають все більш пов'язаними, і може знадобитися встановити додатковий правовий захист.[120]
Перші майже повні секвеновані геноми людини були проведені для двох американців переважно північно-західного європейського походження в 2007 році (Дж. Крейг Вентер із 7,5-кратним охопленням,[121][122][123] і Джеймс Ватсон із 7,4-кратним охопленням).[124][125][126] У 2008 році було проведено секвенування анонімного китайця хань (із 36 кратним охопленням),[127] чоловіка йоруба з Нігерії (із 30 кратним охопленням),[128] жінки-генетика (Марджолейн Крік) з Нідерландів (у 7-8-кратному розмірі) та пацієнтці з лейкемією у віці 50 років (при 33- та 14-кратному охопленні для пухлинних і нормальних тканин).[129] Стів Джобс був серед перших 20 людей, чий геном повністю секвенували, як повідомляється, ціною 100 000 доларів.[130] Станом на червень 2012 було відкрито 69 майже повних геномів людини.[131] У листопаді 2013 року іспанська родина оприлюднила свої особисті геномні дані за ліцензією Creative Commons у вільному доступі. Роботу очолював Мануель Корпас, а дані були отримані шляхом генетичного тестування в організаціях 23andMe та в Пекінськму інституті геноміки . Вважається, що це перший такий набір даних публічних генетичних даних для всієї родини.[132]
Відповідно до журналу Science, основними базами даних повних геномів є:[133]
| Біобанк | Кількість завершених повних секвенованих геномів | Інформація про впуск/доступ |
|---|---|---|
| Біобанк Великобританії | 200 000 | База даних стала доступною через веб-платформу в листопаді 2021 року, і це є найбільший загальнодоступний набір даних цілих геномів.
Ще 300 000 геномів планується оприлюднити на початку 2023 року.[133][134] |
| Trans-Omics для точної медицини | 161 000 | Національний інститут охорони здоров'я США (NIH) вимагає згоди на конкретний проект |
| Програма «Мільйон ветеранів». | 125 000 | Дослідники, які займаються питаннями ветеранів, отримають доступ у 2022 році |
| Genomics England[en]'s 100,000 Genomes[en] | 120 000 | Дослідники повинні долучитися до співпраці |
| All of US | 90 000 | Національний інститут охорони здоров'я США (NIH) планує запустити на початку 2022 року |
З точки зору охоплення генома та точності, повне секвенування генома можна класифікувати за одним із наступних:[135]
- Чорнова послідовність нуклеотидів, що охоплює приблизно 90 % геному з точністю приблизно 99,9 %
- Завершена послідовність нуклеотидів, що охоплює понад 95 % геному з точністю приблизно 99,99 %
Надання справді високоякісної завершеної послідовності нуклеотидів за цим визначенням дуже дороге. Таким чином, більшість результатів «повного секвенування генома» людини є чорновими послідовностями (іноді вищої, а іноді нижчої точності, визначеної вище).[135]
- Coverage (genetics)
- DNA microarray
- DNA profiling
- DNA sequencing
- Duplex sequencing
- Exome Sequencing
- Single cell sequencing
- Horizontal correlation
- Medical genetics
- Nucleic acid sequence
- Human Genome Project
- Personal Genome Project
- Genomics England
- Medical Research Council (MRC)
- Predictive medicine
- Personalized medicine
- Rare functional variant
- SNP annotation
- ↑ Alberts, Bruce; Johnson, Alexander; Lewis, Julian; Raff, Martin; Roberts, Keith; Walter, Peter (2008). Chapter 8. Molecular biology of the cell (вид. 5th). New York: Garland Science. с. 550. ISBN 978-0-8153-4106-2.
- ↑ Definition of whole-genome sequencing – NCI Dictionary of Cancer Terms. National Cancer Institute (англ.). 20 липня 2012. Процитовано 13 жовтня 2018.
- ↑ Gilissen (July 2014). Genome sequencing identifies major causes of severe intellectual disability. Nature. 511 (7509): 344—7. Bibcode:2014Natur.511..344G. doi:10.1038/nature13394. PMID 24896178.
{{cite journal}}:|hdl-access=вимагає|hdl=(довідка) - ↑ Nones, K; Waddell, N; Wayte, N; Patch, AM; Bailey, P; Newell, F; Holmes, O; Fink, JL; Quinn, MC (29 October 2014). Genomic catastrophes frequently arise in esophageal adenocarcinoma and drive tumorigenesis. Nature Communications. 5: 5224. Bibcode:2014NatCo...5.5224N. doi:10.1038/ncomms6224. PMC 4596003. PMID 25351503.
- ↑ van El, CG; Cornel, MC; Borry, P; Hastings, RJ; Fellmann, F; Hodgson, SV; Howard, HC; Cambon-Thomsen, A; Knoppers, BM (June 2013). Whole-genome sequencing in health care. Recommendations of the European Society of Human Genetics. European Journal of Human Genetics. 21 (Suppl 1): S1—5. doi:10.1038/ejhg.2013.46. PMC 3660957. PMID 23819146.
- ↑ Mooney, Sean (Sep 2014). Progress towards the integration of pharmacogenomics in practice. Human Genetics. 134 (5): 459—65. doi:10.1007/s00439-014-1484-7. PMC 4362928. PMID 25238897.
- ↑ Kijk magazine, 01 January 2009
- ↑ Marx, Vivien (11 September 2013). Next-generation sequencing: The genome jigsaw. Nature. 501 (7466): 263—268. Bibcode:2013Natur.501..263M. doi:10.1038/501261a. PMID 24025842.
- ↑ al.], Bruce Alberts ... [et (2008). Molecular biology of the cell (вид. 5th). New York: Garland Science. с. 551. ISBN 978-0-8153-4106-2.
- ↑ Fiers, W.; Contreras, R.; Duerinck, F.; Haegeman, G.; Iserentant, D.; Merregaert, J.; Min Jou, W.; Molemans, F.; Raeymaekers, A. (8 April 1976). Complete nucleotide sequence of bacteriophage MS2 RNA: primary and secondary structure of the replicase gene. Nature. 260 (5551): 500—507. Bibcode:1976Natur.260..500F. doi:10.1038/260500a0. PMID 1264203.
- ↑ Oliver, S. G.; van der Aart, Q. J. M.; Agostoni-Carbone, M. L. та ін. (May 1992). The complete DNA sequence of yeast chromosome III. Nature. 357 (6373): 38—46. Bibcode:1992Natur.357...38O. doi:10.1038/357038a0. PMID 1574125.
- ↑ а б в Fleischmann, R.; Adams, M.; White, O; Clayton, R.; Kirkness, E.; Kerlavage, A.; Bult, C.; Tomb, J.; Dougherty, B. (28 July 1995). Whole-genome random sequencing and assembly of Haemophilus influenzae Rd. Science. 269 (5223): 496—512. Bibcode:1995Sci...269..496F. doi:10.1126/science.7542800. PMID 7542800.
- ↑ Eddy, Sean R. (November 2012). The C-value paradox, junk DNA and ENCODE. Current Biology. 22 (21): R898—R899. Bibcode:2012CBio...22.R898E. doi:10.1016/j.cub.2012.10.002. PMID 23137679.
- ↑ Pellicer, Jaume; FAY, Michael F.; Leitch, Ilia J. (15 September 2010). The largest eukaryotic genome of them all?. Botanical Journal of the Linnean Society. 164 (1): 10—15. doi:10.1111/j.1095-8339.2010.01072.x.
- ↑ Human Genome Sequencing Consortium, International (21 October 2004). Finishing the euchromatic sequence of the human genome. Nature. 431 (7011): 931—945. Bibcode:2004Natur.431..931H. doi:10.1038/nature03001. PMID 15496913.
- ↑ Goffeau, A.; Barrell, B. G.; Bussey, H.; Davis, R. W.; Dujon, B.; Feldmann, H.; Galibert, F.; Hoheisel, J. D.; Jacq, C. (25 October 1996). Life with 6000 Genes. Science. 274 (5287): 546—567. Bibcode:1996Sci...274..546G. doi:10.1126/science.274.5287.546. PMID 8849441. Архів (PDF) оригіналу за 7 March 2016.
- ↑ The C. elegans Sequencing Consortium (11 December 1998). Genome Sequence of the Nematode C. elegans: A Platform for Investigating Biology. Science. 282 (5396): 2012—2018. Bibcode:1998Sci...282.2012.. doi:10.1126/science.282.5396.2012. PMID 9851916.
- ↑ Alberts, Bruce (2008). Molecular Biology of the Cell (вид. 5th). New York: Garland Science. с. 552. ISBN 978-0-8153-4106-2.
- ↑ Dunham, I. (December 1999). The DNA sequence of human chromosome 22. Nature. 402 (6761): 489—495. Bibcode:1999Natur.402..489D. doi:10.1038/990031. PMID 10591208.
- ↑ Adams MD; Celniker SE; Holt RA та ін. (24 березня 2000). The Genome Sequence of Drosophila melanogaster. Science. 287 (5461): 2185—2195. Bibcode:2000Sci...287.2185.. CiteSeerX 10.1.1.549.8639. doi:10.1126/science.287.5461.2185. PMID 10731132.
- ↑ The Arabidopsis Genome Initiative (14 грудня 2000). Analysis of the genome sequence of the flowering plant Arabidopsis thaliana. Nature. 408 (6814): 796—815. Bibcode:2000Natur.408..796T. doi:10.1038/35048692. PMID 11130711.
- ↑ Waterston RH; Lindblad-Toh K; Birney E та ін. (31 жовтня 2002). Initial sequencing and comparative analysis of the mouse genome. Nature. 420 (6915): 520—562. Bibcode:2002Natur.420..520W. doi:10.1038/nature01262. PMID 12466850.
- ↑ International Human Genome Sequencing Consortium (7 вересня 2004). Finishing the euchromatic sequence of the human genome. Nature. 431 (7011): 931—945. Bibcode:2004Natur.431..931H. doi:10.1038/nature03001. PMID 15496913.
- ↑ Braslavsky, Ido та ін. (2003). Sequence information can be obtained from single DNA molecules. Proc Natl Acad Sci USA. 100 (7): 3960—3984. Bibcode:2003PNAS..100.3960B. doi:10.1073/pnas.0230489100. PMC 153030. PMID 12651960.
- ↑ Heger, Monica (2 жовтня 2013). Single-cell Sequencing Makes Strides in the Clinic with Cancer and PGD First Applications. Clinical Sequencing News.
- ↑ Yurkiewicz, I. R.; Korf, B. R.; Lehmann, L. S. (2014). Prenatal whole-genome sequencing--is the quest to know a fetus's future ethical?. New England Journal of Medicine. 370 (3): 195—7. doi:10.1056/NEJMp1215536. PMID 24428465.
- ↑ Staden R (June 1979). A strategy of DNA sequencing employing computer programs. Nucleic Acids Res. 6 (7): 2601—10. doi:10.1093/nar/6.7.2601. PMC 327874. PMID 461197.
- ↑ Edwards, A; Caskey, T (1991). Closure strategies for random DNA sequencing. Methods: A Companion to Methods in Enzymology. 3 (1): 41—47. doi:10.1016/S1046-2023(05)80162-8.
- ↑ Edwards A; Voss H; Rice P; Civitello A; Stegemann J; Schwager C; Zimmermann J; Erfle H; Caskey CT (April 1990). Automated DNA sequencing of the human HPRT locus. Genomics. 6 (4): 593—608. doi:10.1016/0888-7543(90)90493-E. PMID 2341149.
- ↑ Roach JC; Boysen C; Wang K; Hood L (March 1995). Pairwise end sequencing: a unified approach to genomic mapping and sequencing. Genomics. 26 (2): 345—53. doi:10.1016/0888-7543(95)80219-C. PMID 7601461.
- ↑ Fleischmann RD; Adams MD; White O; Clayton RA; Kirkness EF; Kerlavage AR; Bult CJ; Tomb JF; Dougherty BA (July 1995). Whole-genome random sequencing and assembly of Haemophilus influenzae Rd. Science. 269 (5223): 496—512. Bibcode:1995Sci...269..496F. doi:10.1126/science.7542800. PMID 7542800.
{{cite journal}}: Недійсний|displayauthors=29(довідка) - ↑ Adams, MD та ін. (2000). The genome sequence of Drosophila melanogaster. Science. 287 (5461): 2185—95. Bibcode:2000Sci...287.2185.. CiteSeerX 10.1.1.549.8639. doi:10.1126/science.287.5461.2185. PMID 10731132.
- ↑ а б Mukhopadhyay R (February 2009). DNA sequencers: the next generation. Anal. Chem. 81 (5): 1736—40. doi:10.1021/ac802712u. PMID 19193124.
- ↑ Sevim, Volkan; Lee, Juna; Egan, Robert; Clum, Alicia; Hundley, Hope; Lee, Janey; Everroad, R. Craig; Detweiler, Angela M.; Bebout, Brad M. (26 листопада 2019). Shotgun metagenome data of a defined mock community using Oxford Nanopore, PacBio and Illumina technologies. Scientific Data (англ.). 6 (1): 285. Bibcode:2019NatSD...6..285S. doi:10.1038/s41597-019-0287-z. ISSN 2052-4463. PMC 6879543. PMID 31772173.
- ↑ Wang, Yunhao; Zhao, Yue; Bollas, Audrey; Wang, Yuru; Au, Kin Fai (November 2021). Nanopore sequencing technology, bioinformatics and applications. Nature Biotechnology (англ.). 39 (11): 1348—1365. doi:10.1038/s41587-021-01108-x. ISSN 1546-1696. PMC 8988251. PMID 34750572.
- ↑ Definition of whole-genome sequencing – NCI Dictionary of Cancer Terms. National Cancer Institute (англ.). 20 липня 2012. Процитовано 13 жовтня 2018.
- ↑ Strickland, Eliza (14 жовтня 2015). New Genetic Technologies Diagnose Critically Ill Infants Within 26 Hours – IEEE Spectrum. Spectrum.ieee.org. Архів оригіналу за 16 листопада 2015. Процитовано 11 листопада 2016.
- ↑ Article : Race to Cut Whole Genome Sequencing Costs Genetic Engineering & Biotechnology News — Biotechnology from Bench to Business. Genengnews.com. Архів оригіналу за 17 жовтня 2006. Процитовано 23 лютого 2009.
- ↑ Whole Genome Sequencing Costs Continue to Drop. Eyeondna.com. Архів оригіналу за 25 березня 2009. Процитовано 23 лютого 2009.
- ↑ Harmon, Katherine (28 червня 2010). Genome Sequencing for the Rest of Us. Scientific American. Архів оригіналу за 19 березня 2011. Процитовано 13 серпня 2010.
- ↑ San Diego/Orange County Technology News. Sequenom to Develop Third-Generation Nanopore-Based Single Molecule Sequencing Technology. Freshnews.com. Архів оригіналу за 5 грудня 2008. Процитовано 24 лютого 2009.
- ↑ а б Article : Whole Genome Sequencing in 24 Hours Genetic Engineering & Biotechnology News — Biotechnology from Bench to Business. Genengnews.com. Архів оригіналу за 17 жовтня 2006. Процитовано 23 лютого 2009.
- ↑ Pacific Bio lifts the veil on its high-speed genome-sequencing effort. VentureBeat. 10 лютого 2008. Архів оригіналу за 20 лютого 2009. Процитовано 23 лютого 2009.
- ↑ Bio-IT World. Bio-IT World. 6 жовтня 2008. Архів оригіналу за 17 лютого 2009. Процитовано 23 лютого 2009.
- ↑ With New Machine, Helicos Brings Personal Genome Sequencing A Step Closer. Xconomy. 22 квітня 2008. Архів оригіналу за 2 січня 2011. Процитовано 28 січня 2011.
- ↑ Whole genome sequencing costs continue to fall: $300 million in 2003, $1 million 2007, $60,000 now, $5000 by year end. Nextbigfuture.com. 25 березня 2008. Архів оригіналу за 20 грудня 2010. Процитовано 28 січня 2011.
- ↑ Han Cao's nanofluidic chip could cut DNA sequencing costs dramatically. Technology Review. Архів оригіналу за 29 березня 2011.
- ↑ Julia Karow (26 October 2015). BGI Launches Desktop Sequencer in China; Plans to Register Platform With CFDA. GenomeWeb. Процитовано 2 December 2018.
- ↑ BGI Launches New Desktop Sequencer in China, Registers Larger Version With CFDA. 360Dx. GenomeWeb. 11 November 2016. Процитовано 2 December 2018.
- ↑ Monica Heger (26 October 2018). BGI Launches New Sequencer as Customers Report Data From Earlier Instruments. GenomeWeb. Процитовано 2 December 2018.
- ↑ John Carroll (14 липня 2008). Pacific Biosciences gains $100M for sequencing tech. FierceBiotech. Архів оригіналу за 1 травня 2009. Процитовано 23 лютого 2009.
- ↑ Sibley, Lisa (8 лютого 2009). Complete Genomics brings radical reduction in cost. Silicon Valley / San Jose Business Journal. Процитовано 23 лютого 2009.
- ↑ Sarah Neville (5 March 2018). Cheaper DNA sequencing unlocks secrets of rare diseases. Financial Times. Процитовано 2 December 2018.
- ↑ Carlson, Rob (2 січня 2007). A Few Thoughts on Rapid Genome Sequencing and The Archon Prize — synthesis. Synthesis.cc. Архів оригіналу за 8 серпня 2009. Процитовано 23 лютого 2009.
- ↑ «PRIZE Overview: Archon X PRIZE for Genomics».
- ↑ Carlson, Rob (2 січня 2007). A Few Thoughts on Rapid Genome Sequencing and The Archon Prize — synthesis. Synthesis.cc. Архів оригіналу за 8 серпня 2009. Процитовано 23 лютого 2009.
- ↑ «PRIZE Overview: Archon X PRIZE for Genomics».
- ↑ Diamandis, Peter. Outpaced by Innovation: Canceling an XPRIZE. Huffington Post. Архів оригіналу за 25 серпня 2013.
- ↑ Aldhous, Peter. X Prize for genomes cancelled before it begins. Архів оригіналу за 21 вересня 2016.
- ↑ SOLiD System — a next-gen DNA sequencing platform announced. Gizmag.com. 27 жовтня 2007. Архів оригіналу за 19 липня 2008. Процитовано 24 лютого 2009.
- ↑ The $1000 Genome: Coming Soon?. Dddmag.com. 1 квітня 2010. Архів оригіналу за 15 квітня 2011. Процитовано 28 січня 2011.
- ↑ Individual genome sequencing — Illumina, Inc. Everygenome.com. Архів оригіналу за 19 жовтня 2011. Процитовано 28 січня 2011.
- ↑ Illumina launches personal genome sequencing service for $48,000 : Genetic Future. Scienceblogs.com. Архів оригіналу за 16 червня 2009. Процитовано 28 січня 2011.
- ↑ Wade, Nicholas (11 серпня 2009). Cost of Decoding a Genome Is Lowered. The New York Times. Архів оригіналу за 21 травня 2013. Процитовано 3 травня 2010.
- ↑ Technology Index. ABC News. Архів оригіналу за 15 May 2016. Процитовано 29 April 2018.
{{cite web}}: Обслуговування CS1: bot: Сторінки з посиланнями на джерела, де статус оригінального URL невідомий (посилання) - ↑ Drmanac R, Sparks AB, Callow MJ та ін. (2010). Human genome sequencing using unchained base reads on self-assembling DNA nanoarrays. Science. 327 (5961): 78—81. Bibcode:2010Sci...327...78D. doi:10.1126/science.1181498. PMID 19892942.
- ↑ Illumina Announces $5,000 Genome Pricing – Bio-IT World. Архів оригіналу за 17 травня 2011.
- ↑ NHGRI Awards More than $50M for Low-Cost DNA Sequencing Tech Development. Genome Web. 2009. Архів оригіналу за 3 липня 2011.
- ↑ The Cost of Sequencing a Human Genome. www.genome.gov. Архів оригіналу за 25 листопада 2016.
- ↑ With $999 Whole-Genome Sequencing Service, Veritas Embarks on Goal to Democratize DNA Information. 6 March 2016.
- ↑ Andrews, Joe (1 липня 2019). 23andMe competitor Veritas Genetics slashes price of whole genome sequencing 40% to $600. CNBC (англ.). Процитовано 2 вересня 2019.
- ↑ Megan Molteni (18 May 2017). A Chinese Genome Giant Sets Its Sights on the Ultimate Sequencer. Wired. Процитовано 2 December 2018.
- ↑ Phillips, K. A; Pletcher, M. J; Ladabaum, U (2015). Is the "$1000 Genome" Really $1000? Understanding the Full Benefits and Costs of Genomic Sequencing. Technology and Health Care. 23 (3): 373—379. doi:10.3233/THC-150900. PMC 4527943. PMID 25669213.
- ↑ Blog: True Size of a Human Genome | Veritas Genetics. 28 July 2017.
- ↑ Psst, the human genome was never completely sequenced. statnews.com. 20 червня 2017.
- ↑ Genomics Core. Gladstone.ucsf.edu. Архів оригіналу за 30 червня 2010. Процитовано 23 лютого 2009.
- ↑ Nishida N; Koike A; Tajima A; Ogasawara Y; Ishibashi Y; Uehara Y; Inoue I; Tokunaga K (2008). Evaluating the performance of Affymetrix SNP Array 6.0 platform with 400 Japanese individuals. BMC Genomics. 9 (1): 431. doi:10.1186/1471-2164-9-431. PMC 2566316. PMID 18803882.
- ↑ Petrone, Justin (16 January 2007). Illumina, DeCode Build 1M SNP Chip; Q2 Launch to Coincide with Release of Affy's 6.0 SNP Array | BioArray News | Arrays. GenomeWeb. Архів оригіналу за 16 липня 2011. Процитовано 23 лютого 2009.
- ↑ Roach JC; Glusman G; Smit AF та ін. (April 2010). Analysis of genetic inheritance in a family quartet by whole-genome sequencing. Science. 328 (5978): 636—9. Bibcode:2010Sci...328..636R. doi:10.1126/science.1186802. PMC 3037280. PMID 20220176.
- ↑ Campbell CD; Chong JX; Malig M та ін. (November 2012). Estimating the human mutation rate using autozygosity in a founder population. Nat. Genet. 44 (11): 1277—81. doi:10.1038/ng.2418. PMC 3483378. PMID 23001126.
- ↑ Ye K; Beekman M; Lameijer EW; Zhang Y; Moed MH; van den Akker EB; Deelen J; Houwing-Duistermaat JJ; Kremer D (December 2013). Aging as accelerated accumulation of somatic variants: whole-genome sequencing of centenarian and middle-aged monozygotic twin pairs. Twin Res Hum Genet. 16 (6): 1026—32. doi:10.1017/thg.2013.73. PMID 24182360.
- ↑ Keightley PD (February 2012). Rates and fitness consequences of new mutations in humans. Genetics. 190 (2): 295—304. doi:10.1534/genetics.111.134668. PMC 3276617. PMID 22345605.
- ↑ Tuna M; Amos CI (November 2013). Genomic sequencing in cancer. Cancer Lett. 340 (2): 161—70. doi:10.1016/j.canlet.2012.11.004. PMC 3622788. PMID 23178448.
- ↑ Moran, Laurence A. (24 March 2011). Sandwalk: How Big Is the Human Genome?. sandwalk.blogspot.com. Архів оригіналу за 1 December 2017. Процитовано 29 April 2018.
- ↑ Hodgkinson, Alan; Chen, Ying; Eyre-Walker, Adam (January 2012). The large-scale distribution of somatic mutations in cancer genomes. Human Mutation. 33 (1): 136—143. doi:10.1002/humu.21616. PMID 21953857.
- ↑ Supek, Fran; Lehner, Ben (May 2015). Differential DNA mismatch repair underlies mutation rate variation across the human genome. Nature. 521 (7550): 81—84. Bibcode:2015Natur.521...81S. doi:10.1038/nature14173. PMC 4425546. PMID 25707793.
- ↑ Schuster-Böckler, Benjamin; Lehner, Ben (August 2012). Chromatin organization is a major influence on regional mutation rates in human cancer cells. Nature. 488 (7412): 504—507. Bibcode:2012Natur.488..504S. doi:10.1038/nature11273. PMID 22820252.
- ↑ Supek, Fran; Lehner, Ben (July 2017). Clustered Mutation Signatures Reveal that Error-Prone DNA Repair Targets Mutations to Active Genes. Cell. 170 (3): 534—547.e23. doi:10.1016/j.cell.2017.07.003. PMID 28753428.
{{cite journal}}:|hdl-access=вимагає|hdl=(довідка) - ↑ Yano, K; Yamamoto, E; Aya, K; Takeuchi, H; Lo, PC; Hu, L; Yamasaki, M; Yoshida, S; Kitano, H (August 2016). Genome-wide association study using whole-genome sequencing rapidly identifies new genes influencing agronomic traits in rice. Nature Genetics. 48 (8): 927—34. doi:10.1038/ng.3596. PMID 27322545.
- ↑ а б в Borrell, Brendan (14 September 2010). US clinics quietly embrace whole-genome sequencing. Nature: news.2010.465. doi:10.1038/news.2010.465.
- ↑ Ashley, EA; Butte, AJ; Wheeler, MT; Chen, R; Klein, TE; Dewey, FE; Dudley, JT; Ormond, KE; Pavlovic, A (1 May 2010). Clinical assessment incorporating a personal genome. Lancet. 375 (9725): 1525—35. doi:10.1016/S0140-6736(10)60452-7. PMC 2937184. PMID 20435227.
- ↑ Dewey, Frederick E.; Wheeler, Matthew T.; Cordero, Sergio; Perez, Marco V.; Pavlovic, Aleks; Pushkarev, Dmitry; Freeman, James V.; Quake, Steve R.; Ashley, Euan A. (April 2011). Molecular Autopsy for Sudden Cardiac Death Using Whole Genome Sequencing. Journal of the American College of Cardiology. 57 (14): E1159. doi:10.1016/S0735-1097(11)61159-5.
- ↑ Dewey, Frederick E.; Chen, Rong; Cordero, Sergio P.; Ormond, Kelly E.; Caleshu, Colleen; Karczewski, Konrad J.; Whirl-Carrillo, Michelle; Wheeler, Matthew T.; Dudley, Joel T. (15 September 2011). Phased Whole-Genome Genetic Risk in a Family Quartet Using a Major Allele Reference Sequence. PLOS Genetics. 7 (9): e1002280. doi:10.1371/journal.pgen.1002280. PMC 3174201. PMID 21935354.
- ↑ One In A Billion: A boy's life, a medical mystery. Jsonline.com. Архів оригіналу за 5 жовтня 2013. Процитовано 11 листопада 2016.
- ↑ Mayer AN, Dimmock DP, Arca MJ та ін. (March 2011). A timely arrival for genomic medicine. Genet. Med. 13 (3): 195—6. doi:10.1097/GIM.0b013e3182095089. PMID 21169843.
- ↑ Introducing diagnostic applications of '3Gb-testing' in human genetics. Архів оригіналу за 10 листопада 2014.
- ↑ Boccia S, Mc Kee M, Adany R, Boffetta P, Burton H, Cambon-Thomsen A, Cornel MC, Gray M, Jani A, Knoppers BM, Khoury MJ, Meslin EM, Van Duijn CM, Villari P, Zimmern R, Cesario A, Puggina A, Colotto M, Ricciardi W (August 2014). Beyond public health genomics: proposals from an international working group. Eur J Public Health. 24 (6): 877—879. doi:10.1093/eurpub/cku142. PMC 4245010. PMID 25168910.
- ↑ RD-Connect News 18 July 2014. Rd-connect.eu. Архів оригіналу за 10 October 2016. Процитовано 11 листопада 2016.
- ↑ Genomes2People: A Roadmap for Genomic Medicine. www.frontlinegenomics.com. Архів оригіналу за 14 February 2017. Процитовано 29 April 2018.
- ↑ Farnaes, Lauge; Hildreth, Amber; Sweeney, Nathaly M.; Clark, Michelle M.; Chowdhury, Shimul; Nahas, Shareef; Cakici, Julie A.; Benson, Wendy; Kaplan, Robert H. (December 2018). Rapid whole-genome sequencing decreases infant morbidity and cost of hospitalization. npj Genomic Medicine. 3 (1): 10. doi:10.1038/s41525-018-0049-4. PMC 5884823. PMID 29644095.
- ↑ Schwarze, K; Buchanan, J; Taylor, Jc; Wordsworth, S (May 2018). Are whole Exome and whole Genome Sequencing Approaches Cost-Effective? A Systematic Review of the Literature. Value in Health. 21: S100. doi:10.1016/j.jval.2018.04.677.
- ↑ Lee, Seunggeung; Abecasis, Gonçalo R.; Boehnke, Michael; Lin, Xihong (July 2014). Rare-Variant Association Analysis: Study Designs and Statistical Tests. The American Journal of Human Genetics. 95 (1): 5—23. doi:10.1016/j.ajhg.2014.06.009. PMC 4085641. PMID 24995866.
- ↑ Li, Zilin; Li, Xihao; Zhou, Hufeng та ін. (December 2022). A framework for detecting noncoding rare-variant associations of large-scale whole-genome sequencing studies. Nature Methods. 19 (12): 1599—1611. doi:10.1038/s41592-022-01640-x. PMC 10008172. PMID 36303018.
- ↑ STAARpipeline: an all-in-one rare-variant tool for biobank-scale whole-genome sequencing data. Nature Methods. 19 (12): 1532—1533. December 2022. doi:10.1038/s41592-022-01641-w. PMID 36316564.
- ↑ Boston Researchers To Sequence Newborn Babies' DNA. wbur.org. 5 вересня 2013.
- ↑ Holm, Ingrid A.; Agrawal, Pankaj B.; Ceyhan-Birsoy, Ozge; Christensen, Kurt D.; Fayer, Shawn; Frankel, Leslie A.; Genetti, Casie A.; Krier, Joel B.; Lamay, Rebecca C. (9 липня 2018). The BabySeq project: implementing genomic sequencing in newborns. BMC Pediatrics. 18 (1): 225. doi:10.1186/s12887-018-1200-1. PMC 6038274. PMID 29986673.
- ↑ Howard, Heidi Carmen; Knoppers, Bartha Maria; Cornel, Martina C.; Wright Clayton, Ellen; Sénécal, Karine; Borry, Pascal (28 січня 2015). Whole-genome sequencing in newborn screening? A statement on the continued importance of targeted approaches in newborn screening programmes. European Journal of Human Genetics (англ.). 23 (12): 1593—1600. doi:10.1038/ejhg.2014.289. ISSN 1476-5438. PMC 4795188. PMID 25626707.
- ↑ Woerner, Audrey C.; Gallagher, Renata C.; Vockley, Jerry; Adhikari, Aashish N. (19 липня 2021). The Use of Whole Genome and Exome Sequencing for Newborn Screening: Challenges and Opportunities for Population Health. Frontiers in Pediatrics. 9: 663752. doi:10.3389/fped.2021.663752. ISSN 2296-2360. PMC 8326411. PMID 34350142.
- ↑ Tarini, Beth A. (23 серпня 2021). The Effect of BabySeq on Pediatric and Genomic Research—More Than Baby Steps. JAMA Pediatrics. 175 (11): 1107—1108. doi:10.1001/jamapediatrics.2021.2826. PMID 34424259.
- ↑ The Lancet (22 липня 2023). Genomic newborn screening: current concerns and challenges. The Lancet. 402 (10398): 265. doi:10.1016/s0140-6736(23)01513-1. ISSN 0140-6736. PMID 37481265.
- ↑ а б в г д е ж и Sijmons, R.H.; Van Langen, I.M (2011). A clinical perspective on ethical issues in genetic testing. Accountability in Research: Policies and Quality Assurance. 18 (3): 148—162. Bibcode:2013ARPQ...20..143D. doi:10.1080/08989621.2011.575033. PMID 21574071.
- ↑ Ayday E; De Cristofaro E (2015). The Chills and Thrills of Whole Genome Sequencing. arXiv:1306.1264 [cs.CR].
- ↑ Borry, Pascal; Evers-Kiebooms, Gerry; Cornel, Martha C.; Clarke, Angus; Dierickx, Kris (2009). Genetic testing in asymptomatic minors Background considerations towards ESHG Recommendations. European Journal of Human Genetics. 17 (6): 711—9. doi:10.1038/ejhg.2009.25. PMC 2947094. PMID 19277061.
- ↑ Henderson, Mark (9 лютого 2009). Genetic mapping of babies by 2019 will transform preventive medicine. London: Times Online. Архів оригіналу за 11 травня 2009. Процитовано 23 лютого 2009.
- ↑ McCabe LL; McCabe ER (June 2001). Postgenomic medicine. Presymptomatic testing for prediction and prevention. Clin Perinatol. 28 (2): 425—34. doi:10.1016/S0095-5108(05)70094-4. PMID 11499063.
- ↑ Nelson RM; Botkjin JR; Kodish ED та ін. (June 2001). Ethical issues with genetic testing in pediatrics. Pediatrics. 107 (6): 1451—5. doi:10.1542/peds.107.6.1451. PMID 11389275.
- ↑ Borry P; Fryns JP; Schotsmans P; Dierickx K (February 2006). Carrier testing in minors: a systematic review of guidelines and position papers. Eur. J. Hum. Genet. 14 (2): 133—8. doi:10.1038/sj.ejhg.5201509. PMID 16267502.
- ↑ Borry P; Stultiens L; Nys H; Cassiman JJ; Dierickx K (November 2006). Presymptomatic and predictive genetic testing in minors: a systematic review of guidelines and position papers. Clin. Genet. 70 (5): 374—81. doi:10.1111/j.1399-0004.2006.00692.x. PMID 17026616.
- ↑ McGuire, Amy, L; Caulfield, Timothy (2008). Science and Society: Research ethics and the challenge of whole-genome sequencing. Nature Reviews Genetics. 9 (2): 152—156. doi:10.1038/nrg2302. PMC 2225443. PMID 18087293.
- ↑ Curtis, Caitlin; Hereward, James; Mangelsdorf, Marie; Hussey, Karen; Devereux, John (18 December 2018). Protecting trust in medical genetics in the new era of forensics. Genetics in Medicine. 21 (7): 1483—1485. doi:10.1038/s41436-018-0396-7. PMC 6752261. PMID 30559376.
- ↑ Wade, Nicholas (4 вересня 2007). In the Genome Race, the Sequel Is Personal. The New York Times. Архів оригіналу за 11 квітня 2009. Процитовано 22 лютого 2009.
- ↑ Ledford, Heidi (2007). Access : All about Craig: the first 'full' genome sequence. Nature. 449 (7158): 6—7. Bibcode:2007Natur.449....6L. doi:10.1038/449006a. PMID 17805257.
- ↑ Levy S, Sutton G, Ng PC, Feuk L, Halpern AL, Walenz BP, Axelrod N, Huang J, Kirkness EF, Denisov G, Lin Y, MacDonald JR, Pang AW, Shago M, Stockwell TB, Tsiamouri A, Bafna V, Bansal V, Kravitz SA, Busam DA, Beeson KY, McIntosh TC, Remington KA, Abril JF, Gill J, Borman J, Rogers YH, Frazier ME, Scherer SW, Strausberg RL, Venter JC (September 2007). The diploid genome sequence of an individual human. PLOS Biol. 5 (10): e254. doi:10.1371/journal.pbio.0050254. PMC 1964779. PMID 17803354.
- ↑ Wade, Wade (1 червня 2007). DNA pioneer Watson gets own genome map. International Herald Tribune. Архів оригіналу за 27 вересня 2008. Процитовано 22 лютого 2009.
- ↑ Wade, Nicholas (31 травня 2007). Genome of DNA Pioneer Is Deciphered. The New York Times. Архів оригіналу за 20 червня 2011. Процитовано 21 лютого 2009.
- ↑ Wheeler DA; Srinivasan M; Egholm M; Shen Y; Chen L; McGuire A; He W; Chen YJ; Makhijani V (2008). The complete genome of an individual by massively parallel DNA sequencing. Nature. 452 (7189): 872—6. Bibcode:2008Natur.452..872W. doi:10.1038/nature06884. PMID 18421352.
- ↑ Wang J; Wang, Wei; Li, Ruiqiang; Li, Yingrui; Tian, Geng; Goodman, Laurie; Fan, Wei; Zhang, Junqing; Li, Jun (2008). The diploid genome sequence of an Asian individual. Nature. 456 (7218): 60—65. Bibcode:2008Natur.456...60W. doi:10.1038/nature07484. PMC 2716080. PMID 18987735.
{{cite journal}}: Недійсний|displayauthors=29(довідка) - ↑ Bentley DR; Balasubramanian S та ін. (2008). Accurate whole human genome sequencing using reversible terminator chemistry. Nature. 456 (7218): 53—9. Bibcode:2008Natur.456...53B. doi:10.1038/nature07517. PMC 2581791. PMID 18987734.
- ↑ Ley TJ; Mardis ER; Ding L; Fulton B; McLellan MD; Chen K; Dooling D; Dunford-Shore BH; McGrath S (2008). DNA sequencing of a cytogenetically normal acute myeloid leukaemia genome. Nature. 456 (7218): 66—72. Bibcode:2008Natur.456...66L. doi:10.1038/nature07485. PMC 2603574. PMID 18987736.
{{cite journal}}: Недійсний|displayauthors=29(довідка) - ↑ Lohr, Steve (20 жовтня 2011). New Book Details Jobs's Fight Against Cancer. The New York Times. Архів оригіналу за 28 вересня 2017.
- ↑ Complete Human Genome Sequencing Datasets to its Public Genomic Repository. Архів оригіналу за 10 червня 2012.
- ↑ Corpas, Manuel; Cariaso, Mike (12 листопада 2013). A Complete Public Domain Family Genomics Dataset. bioRxiv 10.1101/000216.
- ↑ а б 200,000 whole genomes made available for biomedical studies by U.K. effort. www.science.org (англ.). Процитовано 11 December 2021.
- ↑ Whole Genome Sequencing data on 200,000 UK Biobank participants available now. www.ukbiobank.ac.uk. 17 November 2021. Процитовано 11 December 2021.
- ↑ а б Kris A. Wetterstrand, M.S. The Cost of Sequencing a Human Genome. National Human Genome Research Institute. Last updated: November 1, 2021