Епігенетика
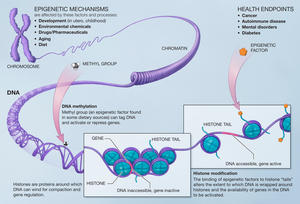

Епігене́тика — наука, галузь біології, яка досліджує зміни в експресії генів або клітинному фенотипі, які відбуваються без змін основної послідовності ДНК. Епігенетика охоплює складний набір молекулярних механізмів, які регулюють те, як гени вмикаються або вимикаються у відповідь на фактори навколишнього середовища, ознаки розвитку та інші внутрішні чи зовнішні стимули.
За своєю суттю епігенетика передбачає модифікації структури ДНК або пов’язаних білків гістонів, що впливають на активність генів. Ці модифікації можуть включати хімічні мітки, такі як метильні групи, додані до молекул ДНК, або зміни білків гістонів, навколо яких намотується ДНК. Ці зміни можуть змінити доступність генів для клітинного механізму, відповідального за зчитування та виконання генетичних інструкцій.
Значення епігенетики полягає в її ролі в контролі різних біологічних процесів, включаючи ембріогенез та розвиток, клітинну диференціацію та сприйнятливість до захворювань. Це підкреслює ідею про те, що наші гени не є лише детермінованими, але можуть залежати від факторів навколишнього середовища, вибору способу життя та досвіду, впливаючи як на індивідуальне здоров’я, так і на успадкування рис між поколіннями.
Вивчення епігенетики має глибоке значення для багатьох наукових дисциплін, включаючи медицину, еволюційну біологію та біологію розвитку. Уявлення, отримані в результаті епігенетичних досліджень, пропонують багатообіцяючі шляхи для розуміння механізмів захворювання, розробки потенційних методів лікування та вивчення взаємодії між генетикою та навколишнім середовищем у формуванні характеристик організму.
Історія[ред. | ред. код]
Історія епігенетики сягає корінням у відкриття метилювання ДНК і модифікацій гістонів.
Дослідження метилювання ДНК[ред. | ред. код]
Про метилювання ДНК вперше було повідомлено в 1940-х і 50-х роках Ролліном Хочкіссом[en] при дослідженні ДНК Escherichia coli.[1][2]
Піонерські дослідження Холлідея[en] та П’ю (1975) заклали основу для розуміння моделей метилювання ДНК та їх ролі в регуляції активності генів.[3] Дослідження Берда (2002)[4] і Єніша[en] та Берда (2003)[5], що з’ясовували вплив метилювання ДНК на мовчання генів, продемонстрували його значення в контролі транскрипції.
Відкриття модифікації гістонів[ред. | ред. код]
Ацетилювання гістонів було відкрито в 1960-х роках Вінсентом Алфрі та його колегами як форму модифікації гістонів, яка може регулювати транскрипцію.[6][7]
Фундаментальне дослідження Алліса[en] та ін. у 2007 році окреслило вплив модифікацій гістонів на структуру хроматину та регуляцію генів, підкресливши динамічну взаємодію між модифікаціями гістонів і транскрипційною активністю.[8]
Дослідження некодуючих РНК[ред. | ред. код]
Дослідження Лі та ін. (1993)[9] і Фаєра та ін. (1998)[10] розкрили ключову роль некодуючих РНК в епігенетичній регуляції, розширюючи розуміння їхньої участі в модуляції експресії генів.
Проект епігенома людини (HEP)[ред. | ред. код]
Започаткований у 2003 році Проєкт Епігенома Людини[en] (Human Epigenome Project) мав на меті скласти карту та зрозуміти епігенетичний ландшафт геному людини, за аналогією Проєкту геному людини. Ініціативи HEP, у тому числі проект «Дорожня карта епігеноміки», створили комплексні епігенетичні карти для різних типів клітин і тканин.
Розвиток епігеномних технологій[ред. | ред. код]
Розвиток високопродуктивних методів секвенування в кінці 2000-х, зробив революцію в епігеномних дослідженнях.[11][12] Ці технології уможливили повногеномний аналіз метилювання ДНК і модифікацій гістонів, значно просунувши сферу епігенетики та епігеноміки.
Молекулярні основи епігенетики[ред. | ред. код]
Епігенетичні модифікації — метилювання ДНК, модифікації гістонів та некодуючі РНК — визначають стани хроматину, впливаючи на доступність ДНК для факторів транскрипції і, таким чином, регулюючи транскрипцію генів та синтез білка.[13]
Метилювання ДНК[ред. | ред. код]
Метилювання ДНК передбачає додавання метильної групи до молекули ДНК, як правило, до залишків цитозину в динуклеотидах CpG, що каталізується ферментами ДНК-метилтрансферазами[14]. Ця модифікація відіграє ключову роль у регуляції експресії генів і структури хроматину. Метильована ДНК часто корелює з мовчанням генів, впливаючи на транскрипційну активність і геномну стабільність.[15][16]
Модифікації гістонів[ред. | ред. код]
Модифікації гістонів охоплюють різноманітний набір хімічних змін, включаючи ацетилювання, метилювання, фосфорилювання та убіквітування, що відбуваються на білках гістонів. Ці модифікації динамічно регулюють структуру хроматину та експресію генів, змінюючи доступність ДНК для механізму транскрипції. Гістонові модифікації діють узгоджено, щоб створити епігенетичний код, диктуючи стан хроматину та активність генів.[17]
Некодуючі РНК[ред. | ред. код]
Некодуючі РНК (нкРНК), включаючи мікроРНК і довгі некодуючі РНК, відіграють вирішальну роль в епігенетичній регуляції.[18] МікроРНК, зазвичай, довжиною 21-23 нуклеотиди, модулюють експресію генів посттранскрипційно, націлюючись на деградацію мРНК або репресію трансляції.[19] Довгі некодуючі РНК, довжина яких перевищує 200 нуклеотидів, регулюють експресію генів на транскрипційному та епігенетичному рівнях, взаємодіючи з комплексами, що модифікують хроматин, і направляючи їх до специфічних геномних локусів.[20]
Структура хроматину та епігенетичний контроль[ред. | ред. код]
Структура хроматину — динамічна збірка ДНК і білків-гістонів — є центральним гравцем епігенетичної регуляції. Організація хроматину в еухроматин (доступний і транскрипційно активний) або гетерохроматин (конденсований і транскрипційно репресований) глибоко впливає на експресію генів. Епігенетичні модифікації — метилювання ДНК, модифікації гістонів та некодуючі РНК — визначають стани хроматину, впливаючи на доступність ДНК і таким чином регулюючи транскрипцію генів.[13]
Епігенетика в біології розвитку[ред. | ред. код]
Основна стаття — Епігенетичне перепрограмування.

Тіло людини містить, за деякими оцінками, 400 основних типів клітин[en][21] — але всі ці типи мають однакову послідовність ДНК — їх відрізняють зміни в епігеномі. Епігеноміка — розділ епігенетики та оміксних аналізів, що передбачає комплексний аналіз і вивчення повного набору епігенетичних модифікацій (тобто, епігенома) у всьому геномі організму.
Епігенетична регуляція ембріонального розвитку[ред. | ред. код]
Епігенетичні модифікації мають вирішальне значення для контролю експресії генів під час ембріогенезу, впливаючи на такі процеси, як клітинна диференціація та органогенез. Ці зміни, які включають метилювання ДНК, модифікації гістонів і механізми на основі РНК, забезпечують правильну просторово-часову експресію генів, уможливлюючи розвиток складних багатоклітинних організмів із однієї заплідненої яйцеклітини.[22][23]
Епігенетика та клітинна диференціація[ред. | ред. код]
На клітинну диференціацію, процес, за допомогою якого клітина змінює один тип на інший, сильно впливають модифікації епігенома. Епігенетичні модифікації можуть «заблокувати» профілі експресії генів диференційованих клітин, гарантуючи, що клітина шкіри, наприклад, продовжує поводитися як клітина шкіри, навіть коли вона ділиться і її нащадки розмножуються.[24]
Епігенетичне перезавантаження в розвитку ссавців[ред. | ред. код]
Унікальною особливістю розвитку ссавців є «епігенетичне перезавантаження», яке відбувається незабаром після запліднення та під час формування статевих клітин. Це передбачає стирання та подальше відновлення епігенетичних позначок, процес, який є критично важливим для підтримки цілісності генома через покоління.[25][26]
Роль некодуючих РНК у розвитку[ред. | ред. код]
Було виявлено, що некодуючі РНК, включаючи мікроРНК і довгі некодуючі РНК, відіграють вирішальну роль у процесах розвитку. Вони можуть модулювати експресію генів на різних рівнях, впливаючи на структуру хроматину, транскрипцію та трансляцію, і тим самим формуючи результати розвитку.[27]
Епігенетика та еволюція[ред. | ред. код]
Епігенетичне успадкування[ред. | ред. код]
Епігенетичні модифікації іноді можуть успадковуватися поколіннями у феномені, відомому як «епігенетичне успадкування[en]».[28] Цей процес, який передбачає передачу інформації від батьків до нащадків, яка не закодована в самій послідовності ДНК, додає ще один рівень складності до нашого розуміння еволюції та природного відбору. Це відкриває розуміння того, що фактори навколишнього середовища, яких зазнає одне покоління, можуть впливати на риси наступних поколінь.[29][30][31]
Епігенетика та видоутворення[ред. | ред. код]
Епігеномні зміни також можуть сприяти процесу видоутворення — утворенню нових і відмінних видів у ході еволюції. Епігенетичні варіації, впливаючи на моделі експресії генів, можуть сприяти фенотипічному різноманіттю, яке спричиняє дивергенцію видів.[32][33]
Епігенетика та адаптація[ред. | ред. код]
Епігеномні модифікації можуть допомогти організмам швидко адаптуватися до змін середовища. На відміну від генетичних мутацій, епігенетичні зміни можуть відбуватися швидко у відповідь на подразники навколишнього середовища, дозволяючи організмам коригувати моделі експресії генів і, отже, свої фенотипи протягом життя.[34]
Епігенетика та еволюційна теорія[ред. | ред. код]
Наслідки епігенетики для еволюції є глибокими, що потенційно вимагає переосмислення традиційної еволюційної теорії. Це передбачає інтеграцію концепції «епігенотипу» в наше розуміння еволюційних процесів поряд із традиційним фокусом на генотипі.[35][36]
Див. також[ред. | ред. код]
- Епігеноміка
- Епігеном
- Епігенетичне перепрограмування
- Епігенетичний ландшафт
- Нутрігеноміка
- Генотерапія
- Омолодження
- Сін'я Яманака
- Редагування епігенома[en]
Додаткова література[ред. | ред. код]
Книги[ред. | ред. код]
- Серга С.В., Козерецька І.А., Демидов С.В., Вайсерман О.М. Епігенетичні основи онтогенезу: Посібник для студентів біологічних, медичних та сільськогосподарських спеціальностей. — Київ.
- Серія книг Books in Epigenomics and Epigenetics (Frontiers in Genetics, 2012-2023+, відкритий доступ)
Журнали[ред. | ред. код]
- Epigenetics
- Clinical Epigenetics
- Epigenetics Communications
- Environmental Epigenetics
- Epigenetics & Chromatin
- Epigenetics Insights
Статті[ред. | ред. код]
- Епігенетика: нескінченна історія (у трьох томах). Том перший. «Моя наука»; Том другий. Власне епігенетика. «Моя наука»
Примітки[ред. | ред. код]
- ↑ Hotchkiss, R. D. (1948-08). The quantitative separation of purines, pyrimidines, and nucleosides by paper chromatography. The Journal of Biological Chemistry. Т. 175, № 1. с. 315—332. ISSN 0021-9258. PMID 18873306. Процитовано 20 червня 2023.
- ↑ Stricker, Stefan H.; Götz, Magdalena (2018). DNA-Methylation: Master or Slave of Neural Fate Decisions?. Frontiers in Neuroscience. Т. 12. doi:10.3389/fnins.2018.00005. ISSN 1662-453X. PMC 5799221. PMID 29449798. Процитовано 10 грудня 2023.
{{cite news}}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання) Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання) - ↑ Holliday, R.; Pugh, J. E. (24 січня 1975). DNA Modification Mechanisms and Gene Activity During Development: Developmental clocks may depend on the enzymic modification of specific bases in repeated DNA sequences. Science (англ.). Т. 187, № 4173. с. 226—232. doi:10.1126/science.187.4173.226. ISSN 0036-8075. Процитовано 9 грудня 2023.
- ↑ Bird, Adrian (1 січня 2002). DNA methylation patterns and epigenetic memory. Genes & Development (англ.). Т. 16, № 1. с. 6—21. doi:10.1101/gad.947102. ISSN 0890-9369. Процитовано 9 грудня 2023.
- ↑ Jaenisch, Rudolf; Bird, Adrian (2003-03). Epigenetic regulation of gene expression: how the genome integrates intrinsic and environmental signals. Nature Genetics (англ.). Т. 33, № 3. с. 245—254. doi:10.1038/ng1089. ISSN 1546-1718. Процитовано 9 грудня 2023.
- ↑ Allfrey, V. G.; Faulkner, R.; Mirsky, A. E. (1964-05). ACETYLATION AND METHYLATION OF HISTONES AND THEIR POSSIBLE ROLE IN THE REGULATION OF RNA SYNTHESIS. Proceedings of the National Academy of Sciences (англ.). Т. 51, № 5. с. 786—794. doi:10.1073/pnas.51.5.786. ISSN 0027-8424. PMC 300163. PMID 14172992. Процитовано 20 червня 2023.
{{cite news}}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання) - ↑ Mukhopadhyay, Rajendrani (2012-01). Vincent Allfrey's Work on Histone Acetylation. Journal of Biological Chemistry. Т. 287, № 3. с. 2270—2271. doi:10.1074/jbc.o112.000248. ISSN 0021-9258. PMC 3265906. Процитовано 10 грудня 2023.
{{cite news}}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання) Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання) - ↑ Allis, C. David; Berger, Shelley L.; Cote, Jacques; Dent, Sharon; Jenuwien, Thomas; Kouzarides, Tony; Pillus, Lorraine; Reinberg, Danny; Shi, Yang (2007-11). New Nomenclature for Chromatin-Modifying Enzymes. Cell (англ.). Т. 131, № 4. с. 633—636. doi:10.1016/j.cell.2007.10.039. Процитовано 9 грудня 2023.
- ↑ Lee, Rosalind C.; Feinbaum, Rhonda L.; Ambros, Victor (1993-12). The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14. Cell. Т. 75, № 5. с. 843—854. doi:10.1016/0092-8674(93)90529-y. ISSN 0092-8674. Процитовано 9 грудня 2023.
- ↑ Fire, Andrew; Xu, SiQun; Montgomery, Mary K.; Kostas, Steven A.; Driver, Samuel E.; Mello, Craig C. (1998-02). Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans. Nature (англ.). Т. 391, № 6669. с. 806—811. doi:10.1038/35888. ISSN 1476-4687. Процитовано 9 грудня 2023.
- ↑ Meissner, Alexander; Mikkelsen, Tarjei S.; Gu, Hongcang; Wernig, Marius; Hanna, Jacob; Sivachenko, Andrey; Zhang, Xiaolan; Bernstein, Bradley E.; Nusbaum, Chad (2008-08). Genome-scale DNA methylation maps of pluripotent and differentiated cells. Nature (англ.). Т. 454, № 7205. с. 766—770. doi:10.1038/nature07107. ISSN 1476-4687. Процитовано 9 грудня 2023.
- ↑ Lister, Ryan; Pelizzola, Mattia; Dowen, Robert H.; Hawkins, R. David; Hon, Gary; Tonti-Filippini, Julian; Nery, Joseph R.; Lee, Leonard; Ye, Zhen (2009-11). Human DNA methylomes at base resolution show widespread epigenomic differences. Nature (англ.). Т. 462, № 7271. с. 315—322. doi:10.1038/nature08514. ISSN 1476-4687. PMC 2857523. PMID 19829295. Процитовано 9 грудня 2023.
{{cite news}}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання) - ↑ а б Handy, Diane E.; Castro, Rita; Loscalzo, Joseph (17 травня 2011). Epigenetic Modifications: Basic Mechanisms and Role in Cardiovascular Disease. Circulation (англ.). Т. 123, № 19. с. 2145—2156. doi:10.1161/CIRCULATIONAHA.110.956839. ISSN 0009-7322. PMC 3107542. PMID 21576679. Процитовано 9 грудня 2023.
{{cite news}}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання) - ↑ Edwards, John R.; Yarychkivska, Olya; Boulard, Mathieu; Bestor, Timothy H. (2017-12). DNA methylation and DNA methyltransferases. Epigenetics & Chromatin (англ.). Т. 10, № 1. doi:10.1186/s13072-017-0130-8. ISSN 1756-8935. PMC 5422929. PMID 28503201. Процитовано 9 грудня 2023.
{{cite news}}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання) Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання) - ↑ Jones, Peter A. (2012-07). Functions of DNA methylation: islands, start sites, gene bodies and beyond. Nature Reviews Genetics (англ.). Т. 13, № 7. с. 484—492. doi:10.1038/nrg3230. ISSN 1471-0064. Процитовано 9 грудня 2023.
- ↑ Moore, Lisa D.; Le, Thuc; Fan, Guoping (2013-01). DNA Methylation and Its Basic Function. Neuropsychopharmacology (англ.). Т. 38, № 1. с. 23—38. doi:10.1038/npp.2012.112. ISSN 1740-634X. Процитовано 9 грудня 2023.
- ↑ Kouzarides, Tony (2007-02). Chromatin Modifications and Their Function. Cell. Т. 128, № 4. с. 693—705. doi:10.1016/j.cell.2007.02.005. ISSN 0092-8674. Процитовано 9 грудня 2023.
- ↑ Wei, Jian-Wei; Huang, Kai; Yang, Chao; Kang, Chun-Sheng (2017-01). Non-coding RNAs as regulators in epigenetics. Oncology Reports (англ.). Т. 37, № 1. с. 3—9. doi:10.3892/or.2016.5236. ISSN 1021-335X. Процитовано 9 грудня 2023.
- ↑ O'Brien, Jacob; Hayder, Heyam; Zayed, Yara; Peng, Chun (2018). Overview of MicroRNA Biogenesis, Mechanisms of Actions, and Circulation. Frontiers in Endocrinology. Т. 9. doi:10.3389/fendo.2018.00402. ISSN 1664-2392. PMC 6085463. PMID 30123182. Процитовано 9 грудня 2023.
{{cite news}}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання) Обслуговування CS1: Сторінки із непозначеним DOI з безкоштовним доступом (посилання) - ↑ Rinn, John L.; Chang, Howard Y. (7 липня 2012). Genome Regulation by Long Noncoding RNAs. Annual Review of Biochemistry (англ.). Т. 81, № 1. с. 145—166. doi:10.1146/annurev-biochem-051410-092902. ISSN 0066-4154. PMC 3858397. PMID 22663078. Процитовано 9 грудня 2023.
{{cite news}}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання) - ↑ Hatton, Ian A.; Galbraith, Eric D.; Merleau, Nono S. C.; Miettinen, Teemu P.; Smith, Benjamin McDonald; Shander, Jeffery A. (26 вересня 2023). The human cell count and size distribution. Proceedings of the National Academy of Sciences (англ.). Т. 120, № 39. doi:10.1073/pnas.2303077120. ISSN 0027-8424. PMC 10523466. PMID 37722043. Процитовано 9 жовтня 2023.
{{cite news}}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання) - ↑ Reik, Wolf (2007-05). Stability and flexibility of epigenetic gene regulation in mammalian development. Nature (англ.). Т. 447, № 7143. с. 425—432. doi:10.1038/nature05918. ISSN 0028-0836. Процитовано 20 червня 2023.
- ↑ Hajkova, Petra (12 серпня 2011). Epigenetic reprogramming in the germline: towards the ground state of the epigenome. Philosophical Transactions of the Royal Society B: Biological Sciences (англ.). Т. 366, № 1575. с. 2266—2273. doi:10.1098/rstb.2011.0042. ISSN 0962-8436. PMC 3130423. PMID 21727132. Процитовано 20 червня 2023.
{{cite news}}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання) - ↑ Mikkelsen, Tarjei S.; Ku, Manching; Jaffe, David B.; Issac, Biju; Lieberman, Erez; Giannoukos, Georgia; Alvarez, Pablo; Brockman, William; Kim, Tae-Kyung (2007-08). Genome-wide maps of chromatin state in pluripotent and lineage-committed cells. Nature (англ.). Т. 448, № 7153. с. 553—560. doi:10.1038/nature06008. ISSN 0028-0836. PMC 2921165. PMID 17603471. Процитовано 20 червня 2023.
{{cite news}}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання) - ↑ Seisenberger, Stefanie; Peat, Julian R; Reik, Wolf (2013-06). Conceptual links between DNA methylation reprogramming in the early embryo and primordial germ cells. Current Opinion in Cell Biology (англ.). Т. 25, № 3. с. 281—288. doi:10.1016/j.ceb.2013.02.013. Процитовано 20 червня 2023.
- ↑ Gruhn, Wolfram H.; Tang, Walfred W.C.; Dietmann, Sabine; Alves-Lopes, João P.; Penfold, Christopher A.; Wong, Frederick C. K.; Ramakrishna, Navin B.; Surani, M. Azim (20 січня 2023). Epigenetic resetting in the human germ line entails histone modification remodeling. Science Advances (англ.). Т. 9, № 3. doi:10.1126/sciadv.ade1257. ISSN 2375-2548. PMC 9848478. PMID 36652508. Процитовано 20 червня 2023.
{{cite news}}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання) - ↑ Guttman, Mitchell; Rinn, John L. (2012-02). Modular regulatory principles of large non-coding RNAs. Nature (англ.). Т. 482, № 7385. с. 339—346. doi:10.1038/nature10887. ISSN 0028-0836. PMC 4197003. PMID 22337053. Процитовано 20 червня 2023.
{{cite news}}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання) - ↑ Jablonka, Eva; Raz, Gal (2009-06). Transgenerational Epigenetic Inheritance: Prevalence, Mechanisms, and Implications for the Study of Heredity and Evolution. The Quarterly Review of Biology (англ.). Т. 84, № 2. с. 131—176. doi:10.1086/598822. ISSN 0033-5770. Процитовано 20 червня 2023.
- ↑ Skinner, Michael K; Guerrero-Bosagna, Carlos; Haque, M Muksitul (3 серпня 2015). Environmentally induced epigenetic transgenerational inheritance of sperm epimutations promote genetic mutations. Epigenetics (англ.). Т. 10, № 8. с. 762—771. doi:10.1080/15592294.2015.1062207. ISSN 1559-2294. PMC 4622673. PMID 26237076. Процитовано 20 червня 2023.
{{cite news}}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання) - ↑ Fitz-James, Maximilian H.; Cavalli, Giacomo (2022-06). Molecular mechanisms of transgenerational epigenetic inheritance. Nature Reviews Genetics (англ.). Т. 23, № 6. с. 325—341. doi:10.1038/s41576-021-00438-5. ISSN 1471-0064. Процитовано 20 червня 2023.
- ↑ Luft, Friedrich C. (4 жовтня 2022). Epigenetic “Transgenerational” Inheritance. Circulation (англ.). Т. 146, № 14. с. 1096—1098. doi:10.1161/CIRCULATIONAHA.122.061794. ISSN 0009-7322. Процитовано 20 червня 2023.
- ↑ Herman, Jacob J.; Spencer, Hamish G.; Donohue, Kathleen; Sultan, Sonia E. (2014-03). HOW STABLE ‘SHOULD’ EPIGENETIC MODIFICATIONS BE? INSIGHTS FROM ADAPTIVE PLASTICITY AND BET HEDGING: SPECIAL SECTION. Evolution (англ.). Т. 68, № 3. с. 632—643. doi:10.1111/evo.12324. Процитовано 20 червня 2023.
- ↑ Greenspoon, Philip B.; Spencer, Hamish G.; M'Gonigle, Leithen K. (2022-06). Epigenetic induction may speed up or slow down speciation with gene flow. Evolution (англ.). Т. 76, № 6. с. 1170—1182. doi:10.1111/evo.14494. ISSN 0014-3820. PMC 9321097. PMID 35482931. Процитовано 20 червня 2023.
{{cite news}}: Обслуговування CS1: Сторінки з PMC з іншим форматом (посилання) - ↑ Angers, Bernard; Castonguay, Emilie; Massicotte, Rachel (2010-04). Environmentally induced phenotypes and DNA methylation: how to deal with unpredictable conditions until the next generation and after. Molecular Ecology (англ.). Т. 19, № 7. с. 1283—1295. doi:10.1111/j.1365-294X.2010.04580.x. Процитовано 20 червня 2023.
- ↑ Bonduriansky, Russell; Day, Troy (1 грудня 2009). Nongenetic Inheritance and Its Evolutionary Implications. Annual Review of Ecology, Evolution, and Systematics (англ.). Т. 40, № 1. с. 103—125. doi:10.1146/annurev.ecolsys.39.110707.173441. ISSN 1543-592X. Процитовано 20 червня 2023.
- ↑ Saunders, Peter T. (2017). Epigenetics and Evolution. Human Development (англ.). Т. 60, № 2-3. с. 81—94. doi:10.1159/000477993. ISSN 0018-716X. Процитовано 20 червня 2023.
| Це незавершена стаття з генетики. Ви можете допомогти проєкту, виправивши або дописавши її. |
| Це незавершена стаття з молекулярної біології. Ви можете допомогти проєкту, виправивши або дописавши її. |
| ||||||||||||||||||||||||||||||||||||||||||||||
| |||||||||||
|