Дезоксирибонуклеїнова кислота: відмінності між версіями
| [неперевірена версія] | [неперевірена версія] |
мНемає опису редагування |
|||
| Рядок 139: | Рядок 139: | ||
== Біологічна роль ДНК == |
== Біологічна роль ДНК == |
||
ДНК є носієм [[генетична інформація|генетичної інформації]], записаною у вигляді послідовності [[нуклеотид]]ів за допомогою [[генетичний код|генетичного коду]]. З молекулами ДНК пов'язано дві основоположні властивості живих організмів — [[спадковість]] і [[мінливість]]. В ході процесу, називаємого [[Реплікація ДНК|реплікацією]] ДНК, утворюються дві копії початкового ланцюжка, що успадковуються дочірніми [[клітина (біологія)|клітинами]] при [[поділ клітини|поділі]]. Клітини, що утворилися, таким чином, будуть генетично ідентичними. Генетична інформація зчитувається при [[екстресія генів|експресії генів]]. В більшості випадків вона використовується для [[біосинтез білків|біосинтезу білків]] у процесах [[Транскрипція (біологія)|транскрипції]] (синтезу молекул [[мРНК]] на матриці ДНК) і [[Трансляція (біологія)|трансляції]] (синтезу [[білок|білків]] на матриці мРНК). |
|||
Послідовність нуклеотідов «кодує» інформацію про різні типи [[РНК]]: кодуючі — [[мРНК|матричні]] або інформаційні (мРНК) та некодуючі — [[рРНК|рибосомальні]] (рРНК), [[тРНК|транспортні]] (тРНК), [[рибозим|каталітичні]] та інші. Всі ці типи РНК синтезуються на основі ДНК в процесі [[транскрипція (біологія)|транскрипції]]. Їх роль в біосинтезі білків та інших процесах життєдіяльності клітини різна. Інформаційна РНК містить інформацію про послідовність [[амінокислота|амінокислот]] в білку, рибосомальні РНК служать основою для [[рибосома|рибосом]] (складних нуклеопротєїнових комплексів, основна функція яких — збірка білку з окремих амінокислот на основі мРНК, транспортні РНК доставляють амінокислоти до місця збірки білків — в активний центр рибосоми, що рухається по мРНК. |
|||
=== Структура геному === |
|||
{{main|Клітинне ядро|Хроматин|Хромосома|Ген}} |
|||
[[Зображення:DNA_Under_electron_microscope_Image_3576B-PH.jpg|thumb|200px|ДНК геному бактеріофага: фотографія під трансмісійним електронним мікроскопом.]] |
|||
ДНК більшості природних [[геном]]ів має дволанцюжкову структуру — або лінійну ([[еукаріоти]], деякі [[вірус]]и і окремі види [[бактерії|бактерій]]), або кільцеву (більшість бактерій та [[археї|архей]], [[хлоропласт]]и і [[мітохондрія|мітохондрії]]). Лінійну одноланцюжкову ДНК містять деякі віруси і [[бактеріофаг]]и. |
|||
У клітинах еукаріотів ДНК розташовується головним чином в [[Клітинне ядро|ядрі]] у вигляді набору [[хромосома|хромосом]]. ДНК бактерій та архей зазвичай представлена однією кільцевою молекулою ДНК, розташованої у вигляді утворення неправильної форми [[цитоплазма|цитоплазмі]], називаємого [[нуклеоїд]]ом <ref>{{cite journal | author = Thanbichler M, Wang S, Shapiro L | title = The bacterial nucleoid: а highly organized and dynamic structure | journal = J Cell Biochem | volume = 96 | issue = 3 | pages = 506 – 21 | year = 2005 | id = PMID 15988757}}</ref>. |
|||
Генетична інформація геному складається з генів. Ген — одиниця передачі спадковій інформації і ділянка ДНК, яка впливає на певну характеристику організму. Ген містить [[відкрита рамка зчитування|відкриту рамку зчитування]], яка транскрибуєтся, а також [[регуляторні послідовності ДНК|регуляторні послідовності]], наприклад, [[Промотор (біологія)|промотор]]и і [[енхансер]]и, які контролюють експресію відкритих рамок зчитування. |
|||
У багатьох [[Вид (біологія)|вид]]ів тільки мала частина загальної послідовності геному кодує білки. Так тільки близько 1,5 % геному людини складається з кодуючих білок [[екзон]]ов, а більше 50 % ДНК людини складається з некодуючих послідовностей ДНК, що повторюються<ref>{{cite journal | author = Wolfsberg T, McEntyre J, Schuler G | title = Guide to the draft human genome | journal = Nature | volume = 409 | issue = 6822 | pages = 824 – 6 | year = 2001 | id = PMID 11236998}}</ref>. Причини наявності такої великої кількості некодуючої ДНК в еукаріотичних геномах і величезна різниця в розмірах геномів ([[C-значення]]) — одна з нерозв'язаних наукових загадок<ref>{{cite journal | author = Gregory T | title = The C-value enigma in plants and animals: а review of parallels and an appeal for partnership | url=http://aob.oxfordjournals.org/cgi/content/full/95/1/133 | journal = Ann Bot (Lond) | volume = 95 | issue = 1 | pages = 133 – 46 | year = 2005}}</ref>. |
|||
=== Послідовності генома, що не кодують білок === |
|||
{{main|Некодуюча ДНК}} |
|||
Традиційно некодуючі послідовності ДНК, за виключенням промоторів, що безпосередньо передують відкритим рамкам зчитування, розглядалися к «сміттєва ДНК» ({{lang-en|junk DNA}}). Проте зараз накопичується все більше даних, що суперечать цій ідеї, і свідчать про різноманітні корисні функції цих послідовностей. [[Теломера|Теломери]] і [[центромера|центромери]] містять мале число генів, але вони важливі для функціонування і стабільності хромосом<ref name=Nugent/><ref>{{cite journal | author = Pidoux A, Allshire R | title = The role of heterochromatin in centromere function | url=http://journals.royalsoc.ac.uk/media/804t6y8vmh5utlb6ua5y/contributions/p/x/7/a/px7ahm740dq5ueuk.pdf | journal = Philos Trans R Soc Lond B Biol Sci | volume = 360 | issue = 1455 | pages = 569 – 79 | year = 2005 | id = PMID 15905142}}</ref>. Форма некодуючих послідовностей людини, що часто зустрічається, — псевдогени, копії генів, інактивовані в результаті [[мутація|мутацій]]<ref>{{cite journal | author = Harrison P, Hegyi H, Balasubramanian S, Luscombe N, Bertone P, Echols N, Johnson T, Gerstein M | title = Molecular fossils in the human genome: identification and analysis of the pseudogenes in chromosomes 21 and 22 | url=http://genome.org/cgi/content/full/12/2/272 | journal = Genome Res | volume = 12 | issue = 2 | pages = 272 – 80 | year = 2002 | id = PMID 11827946}}</ref>. Ці послідовності є чимось ніби молекулярних [[викопні|викопних]], хоча іноді вони можуть служити початковим матеріалом для [[дуплікація генів|дуплікації]] і подальшої [[дивергенція генів|дівергенції генів]]<ref>{{cite journal | author = Harrison P, Gerstein M | title = Studying genomes through the aeons: protein families, pseudogenes and proteome evolution | journal = J Mol Biol | volume = 318 | issue = 5 | pages = 1155 – 74 | year = 2002 | id = PMID 12083509}}</ref>. |
|||
Іший тип некодуючої ДНК, що тим не менш транскрибується в РНК — [[інтрон]]и. Інтрони також є джерелом різноманітності білків в організмі, тому що вони можуть використовуватися як «лінії розрізу і склеювання» при [[сплайсинг|альтернативному сплайсингу]]<ref>{{cite journal | author = Soller M | title = Molecular fossils in the human genome: identification and analysis of the pseudogenes in chromosomes 21 and 22 | url=http://springerlink.com/content/y12529875j170122 | journal = Cell Mol Life Sci | volume = 63 | issue = 7-9 | pages = 796 – 819 | year = 2006 | id = PMID 16465448}}</ref>. Нарешті, некодуючі білок послідовності ДНК можуть кодувати допоміжні клітинні РНК, наприклад [[малі ядерні РНК]]<ref>{{cite journal | author = Michalak P. | title = RNA world - the dark matter of evolutionary genomics| url=http://blackwell-synergy.com/doi/abs/10.1111/j.1420-9101.2006.01141.x | volume = 19 | issue = 6 | pages = 1768 – 74 | year = 2006 | id = PMID 17040373}}</ref>. Недавнє дослідження транскрипції геному людини показало, що 10 % геному дає початок [[поліаденілювання|поліаденільованим]] РНК <ref>{{cite journal | author = Cheng J, Kapranov P, Drenkow J, Dike S, Brubaker S et al. | title = RNA world - the dark matter of evolutionary genomics| url=http://sciencemag.org/cgi/content/full/308/5725/1149 | volume = 308 | pages = 1149 – 54 | year = 2005 | id = PMID 15790807}}</ref>, а дослідження геному миші показало, що 62 % його транскрибується<ref>{{cite journal | author = Mattick JS | title = RNA regulation: а new genetics? | url=http://nature.com/nrg/journal/v5/n4/abs/nrg1321_fs.html;jsessionid=38CA337C2CE6EC04821E4D35AD67995C| journal = Nat Rev Genet| volume = 5 | pages = 316-323 | year = 2004 | id = PMID 15131654}}</ref>. |
|||
=== Транскрипція і трансляція === |
|||
{{main|Генетический код|Транскрипція (біологія)|Трансляція (біологія)}} |
|||
Генетична інформація, закодована в ДНК, повинна бути прочитана і зрештою виражена в синтезі різних [[біополімер]]ів, з яких складаються клітини. Послідовність основ в ланцюжку ДНК безпосередньо визначає послідовність основ в РНК, на яку вона «переписується» в процесі, називаємому транскрипцією. У випадку мРНК, ця послідовність визначає [[амінокислоти]] білку. Співвідношення між нуклеотидною послідовністю мРНК і амінокислотною послідовністю визначається правилами трансляції, які називаються [[генетичний код|генетичним кодом]]. Генетичний код складається із [[кодон]]ів, тринуклеотидних послідовностей (тобто АСТ, САG, ТТТ тощо), що безпосередньо слідують одна за одною. |
|||
Під час транскрипції нуклеотіди гену копіюються на РНК, що синтезується [[РНК-полімераза|РНК-полімеразою]]. Ця копія у разі мРНК декодується [[рибосома|рибосомою]], яка «зчитує» послідовність мРНК, здійснюючи спаровування матричної РНК з ділянками [[тРНК|транспортних РНК]], комплексів РНК і амінокислот. Оскільки в тринуклеотидних комбінаціях використовуються 4 основи, всього можливі 64 кодони (4<sup>3</sup> комбінації). Кодони кодують 20 стандартних амінокислот, кожній з який в більшості випадків відповідає більше за один кодон. Один з трьох кодонів, які розташовуються в кінці мРНК, не кодує амінокислоту і визначає кінець білка, це «стоп» або «нонсенс» кодони (у більшості організмів ТАА, ТGА, ТАG). |
|||
=== Реплікація === |
|||
{{main|Реплікація ДНК}} |
|||
[[Поділ клітини]] необхідний для розмноження [[одноклітинні організми|одноклетинних]] і росту [[багатоклітинні організми|багатоклітинних]] організмів, але до поділу клітина повинна подвоїти геном, щоб дочірні клітини містили ту ж генетичну інформацію, що і початкова клітина. ДНК подвоюється у процесі реплікації, що протікає по [[напівконсервативна реплікація|напівконсервативному]] механізму: два ланцюжки розділяються і потім кожна бракуюча [[Комплементарність|комплементарна]] послідовність ДНК відтворюється ферментом [[ДНК-полимераза|ДНК-полимеразою]]. Цей фермент будує полінуклеотидний ланцюжок, знаходячи правильний нуклеотид через комплементарне спаровування основ і приєднуючи його до ланцюжка, що росте. Основна ДНК-полімераза, що здійснює більшу частину синтезу ([[Голофермент ДНК полімераза III|Pol III]] прокаріотів або Pol δ еукаріотів) не може розпочати синтез нового ланцюжка, а тільки нарощує вже існуючий, тому вона потребує наявності [[праймер]]ів, синтезований за допомогою іншої ДНК полімерази, [[праймаза|праймази]]. Оскільки ДНК-полімерази можуть будувати ланцюжок тільки у напрямі 5' -> 3', для копіювання антипаралельних ланцюгів використовуються складні механізми, що залучають велику кількість ферментів<ref>{{cite journal | author = Albà M | title = Replicative DNA polymerases | url=http://pubmedcentral.nih.gov/articlerender.fcgi?tool=pubmed&pubmedid=11178285 | journal = Genome Biol | volume = 2 | issue = 1 | pages = REVIEWS3002 | year = 2001 | id = PMID 11178285}}</ref>. |
|||
{{Genetics-stub}} |
{{Genetics-stub}} |
||
Версія за 23:57, 9 листопада 2007

Дезоксирибонуклеї́нова кислота́ (ДНК) — один із двох типів нуклеїнових кислот, який забезпечує зберігання, передачу з покоління в покоління і реалізацію генетичної програми розвитку і функціонування живих організмів. Основна роль ДНК в клітинах — довготривале зберігання інформації про структуру РНК і білків.
У клітинах еукаріотів (наприклад, тварин, рослин або грібів) ДНК знаходиться в ядрі клітини в складі хромосом, а також в деяких клітинних органелах (мітохондріях і пластидах). У клітинах прокаріотів (бактерий і архей) кільцева або лінійна молекула ДНК, так званий нуклеоїд, знахидиться в цитоплазмі і прикріплена зсередини до клітинної мембрани. У них і у нижчих еукаріот (наприклад дріжджів) зустрічаються також невеликі автономні кільцеві молекули ДНК, так звані плазміди. Крім того, одно- або дволанцюжкові молекули ДНК можуть утворювати геном ДНК-вірусів.
З хімічної точки зору, ДНК — це довга полімерна молекула, що складається з блоків, що повторюються, нуклеотидів. Кожний нуклеотид складається з азотистої основи, цукру (дезоксирибози) і фосфатної групи. Зв'язки між нуклеотидами в ланцюжку утворюються за рахунок дезоксирибози і фосфатної групи. У переважній більшості випадків (окрім деяких вірусів, що містять одноланцюжкові ДНК) макромолекула ДНК складається з двох ланцюжків, орієнтованих азотистими основами один проти одного. Ця дволанцюжкова молекула утворює спіраль. В цілому структура молекули ДНК отримала назву «подвійної спіралі».
У ДНК зустрічається чотири види азотистих основ (аденін, гуанін, тимін і цитозин) (виняток становлять випадки пізніших модифікацій нуклеотидів, наприклад метилювання). Азотисті основи одного з ланцюжків сполучені з азотистими основами іншого ланцюжка водневими зв'язками згідно принципу комплементарності: аденін з'єднується тільки з тиміном, гуанін — тільки з цитозином. Послідовність нуклеотидів дозволяє «кодувати» інформацію про різні типи РНК, найбільш важливими з яких є інформаційні, або матричні (мРНК), рибосомальні (рРНК) і транспортні (тРНК). Всі ці типи РНК синтезуються на матриці ДНК (тобто за рахунок копіювання послідовності ДНК в послідовність макромолекули, що синтезується) в процесі транскрипції і беруть участь в біосинтезі білків (процесі трансляції). Крім кодуючих послідовностей, ДНК клітин містить послідовності, що виконують регуляторні і структурні функції. Ділянки кодуючої послідосності разом із регуляторними ділянками називаються генами.
У геномах еукаріотів містяться також довгі послідовності без очевидної функції (некодуючі послідовності, інтрони). Також у складі геному досить поширені генетичні паразити — транспозони і вірусні або схожі на них послідовності.
Розшифровка структури ДНК (виконана в 1953 році) стала одним з поворотних моментів в історії біології. За видатний внесок в це відкриття Френсісу Кріку, Джеймсу Ватсону і Морісу Вілкінсу була присуджена Нобелівська премія з фізіології і медицини 1962 року.
Історія дослідження

ДНК була відкрита Іоганном Фрідріхом Мішером в 1869 році. Спочатку нова речовина отримала назву нуклєїн, а пізніше, коли Мішер визначив, що ця речовина володіє кислотними властивостями, речовина отримала назву нуклеїнова кислота[1]. Біологічна функція нововідкритої речовини була неясна, і довгий час ДНК вважалася запасником фосфору в організмі. Більш того, навіть на початку 20 століття багато біологів вважали, що ДНК не має ніякого відношення до передачі інформації, оскільки будова молекули, на їх думку, була дуже одноманітною і не могла містити закодовану інформацію.
Поступово було доведено, що саме ДНК, а не білки, як вважалося раніше, є носієм генетичної інформації. Один з перших вирішальних доказів стали експерименти О. Евері, Коліна Мак-Леода і Маклін Мак-Карті (1944 рік) по трансформації бактерій. Їм вдалося показати, що за так звану трансформацію (придбання хвороботворних властивостей нешкідливою культурою в результаті додавання в неї мертвих хвороботворних бактерій) відповідає виділена з пневмококів ДНК. Експеримент американських учених Алфреда Хершу і Марти Чейз (1952 рік) з поміченими радіоактивними ізотопами білками і ДНК бактеріофагів показали, що в заражену клітку передається тільки нуклеїнова кислота фага, а нове покоління фага містить такі ж білки і нуклеїнову кислоту, як і початковий фаг[2].
До 50-х років 20 століття точна будова ДНК, як і спосіб передачі спадковій інформації, залишалася невідомою. Хоч і було напевно відомо, що ДНК складається з кількох ланцюжків, що у свою чергу складаються з нуклеотидів, ніхто не знав точно, скільки цих ланцюжків і як вони сполучені.
Структура подвійної спіралі ДНК була запропонована Френсісом Кріком і Джеймсом Ватсоном в 1953 році на основі рентгеноструктурних даних, отриманих Морісом Вілкінсом і Розаліндой Франклін, і «правил Чаргаффа», згідно яким в кожній молекулі ДНК дотримуються строгі співвідношення, що зв'язують між собою кількість азотистих основ різних типів[3]. Пізніше запропонована Ватсоном і Кріком модель будови ДНК була доведена, а їх робота відмічена Нобелевскою премією з фізіології і медицини 1962 року. Серед одержувачів не було Розалінди Франклін, що померла на той час, оскільки премія не присуджується посмертно[4].
У відомій доповіді 1957 року, Крік окреслив основи так званої «Центральної догми» молекулярної біології, яка передбачає взаємовідношення між ДНК, РНК, і білками, і сформулював «адаптерну гіпотезу»[5]. Остаточне підтвердження механізму копіювання, запропонованого на основі спіральної структури, було отримане в 1958 році за допомогою експерименту Мезельсона-Сталя[6]. Подальші роботи Кріка і його лабораторії показали, що генетичний код засновується на трійках основ, що не перекриваються, кодонах, що пізніше дозволило Гару Ґобінду Хорані, Роберту Голлі і Маршаллу Ніренбергу розшифрувати генетичний код[7]. Ці відкриття позначають початок молекулярної біології.
Структура молекули
Нуклеотиди

|

|
|

|

|
| Аденін | Гуанін | Тимін | Цитозин | Аденозинмонофосфат |
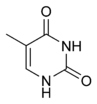
Дезоксирибонуклеїнова кислота є біополімером (поліаніоном), мономерами якого є нуклеотиди[8][9]. Кожен нуклеотид складається із залишку фосфорної кислоти, приєднаного по 5'-положенню до цукру дезоксирибози, до якого також через глікозидний зв'язок (C—N) по 1'-положенню приєднана одна з чотирьох азотистих основ. Саме наявність характерного цукру і складає одну з головних відмінностей між ДНК і РНК, зафіксовану в назвах цих нуклеїнових кислот (до складу РНК входить цукор рибоза)[10]. Приклад нуклеотиду — аденозинмонофосфат — де основа, приєднана до фосфату і рибози, це аденін, показаний на малюнку.
Виходячи із структури молекул, основи, що входять до складу нуклеотидів, розділяють на дві групи: пуринові (аденін [A] і гуанін [G]), утворені сполученими п'яти- і шістичленним гетероциклами і піримідинові (цитозин [C] і тимін [T]) — утворені одним шестичленним гетероциклом[11].
Як виняток, наприклад, у бактеріофага PBS1, в ДНК зустрічається п'ятий тип основ — урацил ([U]), піримідінова основа, що зазвичай входить до складу РНК замість тиміну і відрізняється від тиміну відсутністю метильної групи на кільці[12]. Слід зазначити, що тимін і урацил не так строго приурочені до ДНК і РНК відповідно, як це вважалося раніше. Так, після синтезу деяких молекул РНК значне число урацилов в цих молекулах метилюєтся за допомогою спеціальних ферментів, перетворюючись на тимін. Це відбувається в транспортних і рибосомних РНК[13].
Подвійна спіраль

Полімер ДНК має досить складну структуру. Нуклеотиди ковалентно сполучені між собою в довгі полінуклеотидні ланцюжки. Ці ланцюжки в переважній більшості випадків (окрім деяких вірусів, що мають одноланцюжковий ДНК-геном), у свою чергу, попарно об'єднуються за допомогою водневих зв'язків в структуру, що отримала назву подвійної спіралі[3][10]. Фосфатні групи формують фосфодіефірні зв'язки між третім і п'ятим атомами вуглецю сусідніх молекул дезоксирибози, в результаті взаємодії між 3-гідроксильною (3 —ОН) групою однієї молекули дезоксирибози і 5-фосфатною групою (5 —РО3) іншої. Асиметричні кінці ланцюжка ДНК називаються 3' (три-прайм) і 5' (п'ять-прайм). Полярність ланцюжка грає важливу роль при синтезі ДНК (подовження ланцюжка можливе тільки шляхом приєднання нових нуклеотидів до вільного 3' кінцю).
Як вже було сказано вище, у переважної більшості живих організмів ДНК складається не з одного, а з двох полінуклеотидних ланцюжків. Ці два довгі ланцюжки закручені один навколо іншого у вигляді подвійної спіралі, що стабілізується водневими зв'язками, які утворюються між повернутими один до одного азотистими основами ланцюжків, що входять до неї. У природі ця спіраль, зазвичай, правозакручена. Напрями від 3' кінця до 5' кінця в двох ланцюжках, з яких складається молекула ДНК, протилежні (ланцюжки «антипаралельні» один одному).
Ширина подвійної спіралі в її найбільш поширеній B-формі складає від 22 до 24 Å, або 2,2 — 2,4 нм, а довжина кожного нуклеотида 3,3 Å (0,33 нм)[15]. Подібно до того, як в гвинтових сходах збоку можна побачити сходинки, на подвійній спіралі ДНК в проміжках між фосфатним остовом молекули можна бачити ребра основ, кільця яких розташовані в площині, перпендикулярній по відношенню до подовжньої осі макромолекули.
У подвійній спіралі розрізняють малу (12 Å) і велику (22 Å) бороздки[16]. Білки, наприклад, фактори транскрипції, які приєднуються до певних послідовностей в дволанцюжковій ДНК, зазвичай взаємодіють з краями основ у великій бороздці, де вони доступніші[17].
Утворення зв'язків між основами
Кожна основа на одному з ланцюжків зв'язується з однією певною основою на іншому ланцюжку. Таке специфічне зв'язування називається комплементарним. Пуринові основи комплементарні піримідиновим (тобто, здібні до утворення водневих зв'язків з ними): аденін утворює зв'язки тільки з тиміном, а цитозин — з гуаніном. У подвійній спіралі ланцюжки також зв'язані за допомогою гідрофобної взаємодії і стекінгу, які не залежать від послідовності основ ДНК[18].
Комплементарність подвійної спіралі означає, що інформація, що міститься в одному ланцюжку, міститься і в іншому ланцюжку. Оборотність і специфічність взаємодій між комплементарними парами основ важлива для реплікації ДНК і решти всіх функцій ДНК в живих організмах.
Оскільки водневі зв'язки нековалентні, вони легко разриваюся і відновлюються. Ланцюжки подвійної спіралі можуть розходитися як замок-змійка під дією ферментів (гелікази) або при високій температурі[19].
Різні пари основ утворюють різну кількість водневих зв'язків. A-T зв'язані двома, G-C — трьома водневими зв'язками, тому на розрив GC потрібно більше енергії. Відсоток GC пар і довжина молекули ДНК визначають кількість енергії, необхідної для дисоціації ланцюжків: довгі молекули ДНК з великим вмістом GC більш «туготопкі»[20].
Альтернативні форми подвійної спіралі

ДНК може існувати в кількох можливих конформаціях. Зараз ідентифіковані та описані наступні: A-ДНК, B-ДНК, C-ДНК, D-ДНК[21], E-ДНК[22], H-ДНК[23], L-ДНК[21], P-ДНК[24] і Z-ДНК[25][26]. Проте, тільки A-, B- і Z- форми ДНК спостерігалися в природних біологічних системах. Конформація, яку приймає ДНК, залежить від послідовності ДНК, величини та напрямку суперскрученості, хімічних модифікації основ і концентрації хімічних речовин в розчині, перш за все концентрацій іонів металів і поліамінів[27]. Із всіх конформацій, B-форма, описана вище, є найзагальнішою формою за умовами, знайденими у більшості клітин[28]. Альтернативні конформації подвійної спіралі відрізняються своєю геомертією та розмірами.
A-форма — ширша правостороння спіраль, з дрібнішою і ширшою малою бороздкою і вужчою і глибшою великою бороздкою. Ця форма зустрічається за нефізіологічними умовами в зневоднених зразках ДНК, крім того вона ймовірно зустрічається в живих клітинах в гібридних комплексах ланцюжків ДНК і РНК, та в комплексах ферментної ДНК[29][30]. Сегменти ДНК із хімічно зміненими (метильованими) основами можуть проходити через більші конформаційні зміни і приймають Z-форму. Тут, ланцюжки закручаються в ліву подвійну спіраль, на відміну від правої спіралі B-форми[31]. Ці структури можуть розпознаватися специфічними Z-ДНК зв'язуючими білками і можуть бути залучені в регуляцію транскріпції[32].
Суперскрученість
Якщо узятися за кінці мотузки і почати скручувати їх в різні боки, вона стає коротшою і на мотузкі утворюються великі «супервитки». Також може бути суперскручена і ДНК. У звичайному стані ланцюжок ДНК робить один оборот на кожні 10,4 основ, але в суперскрученому стані спіраль може бути згорнута тугіше або розплетена[33]. Виділяють два типи суперскрученості: позитивну — у напрямі нормальних витків, при якому основи розташовані ближче одна до одної; і негативне — в протилежному напрямку. У природі молекули ДНК зазвичай знаходяться в стані негативної суперскрученості, який вноситься ферментами, — топоізомеразами[34]. Ці ферменти вилучають додаткову скрученість, що виникає в ДНК в результате транскрипції и реплікації[35].
Структури на кінцях хромосом

На кінцях лінійних хромосом знаходяться спеціалізовані структури ДНК, називаємі теломерами. Основна функція цих ділянок — підтримка цілісності кінців хромосом[37]. Оскільки звичайна ДНК-полімераза не може реплікувати 3'-кінці хромосом, спеціальний фермент — теломераза — після кожного поділу клітини додає до хромосом повторювані ділянки ДНК, теломери.
Теломери також захищають кінці ДНК від деградації екзонуклеазами і запобігають активації системи репарації, які запускаються у відповідь на розриви ДНК[38].
У клітках людини теломери зазвичай представлені одноланцюжковою ДНК і складаються з декілька тисяч одиниць послідовності TTAGGG[39]. Ці послідовності з високим вмістом гуаніну стабілізують кінці хромосом, формуючи дуже незвичайні структури, називаємі G-квадруплексами і що складаються з чотирьох, а не двох взаємодіючих основ. Чотири гуанінових основи, всі атоми яких знаходяться в одній площині, утворюють пластинку, стабілізовану водневими зв'язками між основами і хелатуванням в центрі її іона металу (найчастіше калію). Ці пластинки складаються стопкою друг над другом[40].
На кінцях хромосом можуть утворюватися і інші структури: основи можуть бути розташовані в одному ланцюжку або в різних паралельних ланцюжках. Окрім цих структур «стопок», теломери формують великі пітлеподібні структури, звані Т-пітлі або теломерні пітли. У них одноланцюжкова ДНК розташовується у вигляді широкого кільця, стабілізованого теломерними білками[41]. В кінці Т-петлі одноланцюжкова теломерна ДНК приєднується до двуланцюжкової ДНК, порушуючи спаровування ланцюжків в цій молекулі і утворюючи зв'язки з одним з ланцюжків. Це триланцюжкове утворення називається D-пітлею (від англ. displacement loop)[40].
Хімічні модифікації
Метилювання ДНК

|

|

|
| Цитозин | 5-метилцитозин | тимін |
За певними умовами основи ДНК підвергаються хімічним модифікаціям, які можуть бути успадковані без заміни послідовності ДНК, і, таким чином, є частиною епігенетичного коду. Найпоширенішим і найкраще описаних механізмом хімічних модифікацій є метиювання основ ДНК. Метилювання ДНК знайдене у всіх клітинах, проте середній рівень метилювання відрізняється у різних організмів, так у нематоди Caenorhabditis elegans метилювання цитозіну майже не спостерігається, а у хребетних виявлений високий рівень метилювання — до 1 % [42].
Відомо, що рівень метилювання цитозину впливає на експресію генів: ділянки гетерохроматіну (що характеризуються відсутністю або низьким рівнем транскрипції) корелюють із рівнем метилювання. Наприклад, метилювання цитозіну з утворенням 5'-метилцитозину важливе для інактивації Х-хромосоми[43].
Не дивлячись на біологічну роль, 5-метилцитозин може спонтанно деамінуватися, перетворюючись на тимін, тому метильовані цитозини є джерелом підвищеного числа мутаций[44].
Інші модифікації основ включають метилювання аденіну у бактерій і глікозування урацила з утворенням «J-основи» в кінетопластах[45].
Пошкодження ДНК

ДНК може пошкоджуватись різноманітними мутагенами, до яких відносяться окислюючі і алкілюючі речовини, а також високоенергетична магнітна радіація — ультрафіолетове і рентгенівське випромінювання. Тип пошкодження ДНК залежить від тип мутагена. Наприклад, ультрафіолет пошкоджує ДНК шляхом появи в ній димерів тиміну, які утворюються при формуванні ковалентних зв'язків між сусідніми основами[47].
Активні форми кисню, наприклад «вільні» радикали або перекис водню призводять до кількох типів пошкодження ДНК, включаючи модифікації основ, особливо гуанозину, а також дволанцюжкові розриви в ДНК[48]. За деякими оцінками у кожній клітині людини порядку 500 основ пошкоджуються окислюючими сполуками кожного дня[49][50]. Серед різних типів пошкоджень найбільш небезпечні — це дволанцюжкові розриви, тому що вони важко репаруються і можуть призвести до втрат ділянок хромосом (делецій) і транслокацій.
Багато молекул мутагенів вставляються (інтеркалюють) між двома сусідніми парами основ. Більшість цих сполук, наприклад, бромістий етидій, дауноміцин, доксорубіцин і талідомід мають ароматичну структуру. Для того, щоби ароматична сполука могла вміститися між основами, вони повинні розійтися, розплітаючи і порушуючи структуру подвійної спіралі. Ці зміни в структурі ДНК перешкоджують транскрипції і реплікації, викликаючи мутації. Тому інтеркалюючі речовини часто є канцерогенами, найвідоміші з яких — бензопірен, акридини, афлатоксин і бромістий етидій[51][52][53]. Незважаючи на ці негативні властивості, в силу їх здатності пригнічувати транскрипцію і реплікацію ДНК, ці речовини використовуються в хіміотерапії для пригнічення швидкого росту ракових клітин[54].
Біологічна роль ДНК
ДНК є носієм генетичної інформації, записаною у вигляді послідовності нуклеотидів за допомогою генетичного коду. З молекулами ДНК пов'язано дві основоположні властивості живих організмів — спадковість і мінливість. В ході процесу, називаємого реплікацією ДНК, утворюються дві копії початкового ланцюжка, що успадковуються дочірніми клітинами при поділі. Клітини, що утворилися, таким чином, будуть генетично ідентичними. Генетична інформація зчитувається при експресії генів. В більшості випадків вона використовується для біосинтезу білків у процесах транскрипції (синтезу молекул мРНК на матриці ДНК) і трансляції (синтезу білків на матриці мРНК).
Послідовність нуклеотідов «кодує» інформацію про різні типи РНК: кодуючі — матричні або інформаційні (мРНК) та некодуючі — рибосомальні (рРНК), транспортні (тРНК), каталітичні та інші. Всі ці типи РНК синтезуються на основі ДНК в процесі транскрипції. Їх роль в біосинтезі білків та інших процесах життєдіяльності клітини різна. Інформаційна РНК містить інформацію про послідовність амінокислот в білку, рибосомальні РНК служать основою для рибосом (складних нуклеопротєїнових комплексів, основна функція яких — збірка білку з окремих амінокислот на основі мРНК, транспортні РНК доставляють амінокислоти до місця збірки білків — в активний центр рибосоми, що рухається по мРНК.
Структура геному

ДНК більшості природних геномів має дволанцюжкову структуру — або лінійну (еукаріоти, деякі віруси і окремі види бактерій), або кільцеву (більшість бактерій та архей, хлоропласти і мітохондрії). Лінійну одноланцюжкову ДНК містять деякі віруси і бактеріофаги.
У клітинах еукаріотів ДНК розташовується головним чином в ядрі у вигляді набору хромосом. ДНК бактерій та архей зазвичай представлена однією кільцевою молекулою ДНК, розташованої у вигляді утворення неправильної форми цитоплазмі, називаємого нуклеоїдом [55].
Генетична інформація геному складається з генів. Ген — одиниця передачі спадковій інформації і ділянка ДНК, яка впливає на певну характеристику організму. Ген містить відкриту рамку зчитування, яка транскрибуєтся, а також регуляторні послідовності, наприклад, промотори і енхансери, які контролюють експресію відкритих рамок зчитування.
У багатьох видів тільки мала частина загальної послідовності геному кодує білки. Так тільки близько 1,5 % геному людини складається з кодуючих білок екзонов, а більше 50 % ДНК людини складається з некодуючих послідовностей ДНК, що повторюються[56]. Причини наявності такої великої кількості некодуючої ДНК в еукаріотичних геномах і величезна різниця в розмірах геномів (C-значення) — одна з нерозв'язаних наукових загадок[57].
Послідовності генома, що не кодують білок
Традиційно некодуючі послідовності ДНК, за виключенням промоторів, що безпосередньо передують відкритим рамкам зчитування, розглядалися к «сміттєва ДНК» (англ. junk DNA). Проте зараз накопичується все більше даних, що суперечать цій ідеї, і свідчать про різноманітні корисні функції цих послідовностей. Теломери і центромери містять мале число генів, але вони важливі для функціонування і стабільності хромосом[38][58]. Форма некодуючих послідовностей людини, що часто зустрічається, — псевдогени, копії генів, інактивовані в результаті мутацій[59]. Ці послідовності є чимось ніби молекулярних викопних, хоча іноді вони можуть служити початковим матеріалом для дуплікації і подальшої дівергенції генів[60].
Іший тип некодуючої ДНК, що тим не менш транскрибується в РНК — інтрони. Інтрони також є джерелом різноманітності білків в організмі, тому що вони можуть використовуватися як «лінії розрізу і склеювання» при альтернативному сплайсингу[61]. Нарешті, некодуючі білок послідовності ДНК можуть кодувати допоміжні клітинні РНК, наприклад малі ядерні РНК[62]. Недавнє дослідження транскрипції геному людини показало, що 10 % геному дає початок поліаденільованим РНК [63], а дослідження геному миші показало, що 62 % його транскрибується[64].
Транскрипція і трансляція
Генетична інформація, закодована в ДНК, повинна бути прочитана і зрештою виражена в синтезі різних біополімерів, з яких складаються клітини. Послідовність основ в ланцюжку ДНК безпосередньо визначає послідовність основ в РНК, на яку вона «переписується» в процесі, називаємому транскрипцією. У випадку мРНК, ця послідовність визначає амінокислоти білку. Співвідношення між нуклеотидною послідовністю мРНК і амінокислотною послідовністю визначається правилами трансляції, які називаються генетичним кодом. Генетичний код складається із кодонів, тринуклеотидних послідовностей (тобто АСТ, САG, ТТТ тощо), що безпосередньо слідують одна за одною.
Під час транскрипції нуклеотіди гену копіюються на РНК, що синтезується РНК-полімеразою. Ця копія у разі мРНК декодується рибосомою, яка «зчитує» послідовність мРНК, здійснюючи спаровування матричної РНК з ділянками транспортних РНК, комплексів РНК і амінокислот. Оскільки в тринуклеотидних комбінаціях використовуються 4 основи, всього можливі 64 кодони (43 комбінації). Кодони кодують 20 стандартних амінокислот, кожній з який в більшості випадків відповідає більше за один кодон. Один з трьох кодонів, які розташовуються в кінці мРНК, не кодує амінокислоту і визначає кінець білка, це «стоп» або «нонсенс» кодони (у більшості організмів ТАА, ТGА, ТАG).
Реплікація
Поділ клітини необхідний для розмноження одноклетинних і росту багатоклітинних організмів, але до поділу клітина повинна подвоїти геном, щоб дочірні клітини містили ту ж генетичну інформацію, що і початкова клітина. ДНК подвоюється у процесі реплікації, що протікає по напівконсервативному механізму: два ланцюжки розділяються і потім кожна бракуюча комплементарна послідовність ДНК відтворюється ферментом ДНК-полимеразою. Цей фермент будує полінуклеотидний ланцюжок, знаходячи правильний нуклеотид через комплементарне спаровування основ і приєднуючи його до ланцюжка, що росте. Основна ДНК-полімераза, що здійснює більшу частину синтезу (Pol III прокаріотів або Pol δ еукаріотів) не може розпочати синтез нового ланцюжка, а тільки нарощує вже існуючий, тому вона потребує наявності праймерів, синтезований за допомогою іншої ДНК полімерази, праймази. Оскільки ДНК-полімерази можуть будувати ланцюжок тільки у напрямі 5' -> 3', для копіювання антипаралельних ланцюгів використовуються складні механізми, що залучають велику кількість ферментів[65].
| Це незавершена стаття з генетики. Ви можете допомогти проєкту, виправивши або дописавши її. |
Шаблон:Link FA Шаблон:Link FA Шаблон:Link FA Шаблон:Link FA Шаблон:Link FA
- ↑ Dahm R (2005). Friedrich Miescher and the discovery of DNA. Dev Biol. 278 (2): 274—88. PMID 15680349.
- ↑ Hershey A, Chase M (1952). Independent functions of viral protein and nucleic acid in growth of bacteriophage (PDF). J Gen Physiol. 36 (1): 39—56. PMID 12981234.
- ↑ а б Watson J, Crick F (1953). Molecular structure of nucleic acids; a structure for deoxyribose nucleic acid (PDF). Nature. 171 (4356): 737—8. PMID 13054692.
- ↑ The Nobel Prize in Physiology or Medicine 1962 Nobelprize .org Accessed 22 Dec 06
- ↑ Crick, F.H.C. On degenerate templates and the adaptor hypothesis (PDF). genome.wellcome.ac.uk (Lecture, 1955). Accessed 22 Dec 2006
- ↑ Meselson M, Stahl F (1958). The replication of DNA in Escherichia coli. Proc Natl Acad Sci U S A. 44 (7): 671—82. PMID 16590258.
- ↑ The Nobel Prize in Physiology or Medicine 1968 Nobelprize.org Accessed 22 Dec 06
- ↑ Alberts, Bruce; Alexander Johnson, Julian Lewis, Martin Raff, Keith Roberts, and Peter Walters (2002). Molecular Biology of the Cell; Fourth Edition. New York and London: Garland Science. ISBN 0-8153-3218-1.
- ↑ Butler, John M. (2001) Forensic DNA Typing «Elsevier». pp. 14 — 15. ISBN 978-0-12-147951-0
- ↑ а б Berg J., Tymoczko J. and Stryer L. (2002) Biochemistry. W. H. Freeman and Company ISBN 0-7167-4955-6
- ↑ Abbreviations and Symbols for Nucleic Acids, Polynucleotides and their Constituents IUPAC-IUB Commission on Biochemical Nomenclature (CBN) Accessed 03 Jan 2006
- ↑ Takahashi I, Marmur J. (1963). Replacement of thymidylic acid by deoxyuridylic acid in the deoxyribonucleic acid of а transducing phage for Bacillus subtilis. Nature. 197: 794—5. PMID 13980287.
- ↑ Agris P (2004). Decoding the genome: а modified view. Nucleic Acids Res. 32 (1): 223—38. PMID 14715921.
- ↑ Зроблено за даними PDB 1D65
- ↑ Mandelkern M, Elias J, Eden D, Crothers D (1981). The dimensions of DNA in solution. J Mol Biol. 152 (1): 153—61. PMID 7338906.
- ↑ Wing R, Drew H, Takano T, Broka C, Tanaka S, Itakura K, Dickerson R (1980). Crystal structure analysis of а complete turn of B-DNA. Nature. 287 (5784): 755—8. PMID 7432492.
- ↑ Pabo C, Sauer R. PROTEIN-DNA recognition. Annu Rev Biochem. 53: 293—321. PMID 6236744.
- ↑ Ponnuswamy P, Gromiha M (1994). On the conformational stability of oligonucleotide duplexes and tRNA molecules. J Theor Biol. 169 (4): 419—32. PMID 7526075.
- ↑ Clausen-Schaumann H, Rief M, Tolksdorf C, Gaub H (2000). Mechanical stability of single DNA molecules. Biophys J. 78 (4): 1997—2007. PMID 10733978.
- ↑ Chalikian T, Völker J, Plum G, Breslauer K (1999). A more unified picture for the thermodynamics of nucleic acid duplex melting: а characterization by calorimetric and volumetric techniques. Proc Natl Acad Sci U S A. 96 (14): 7853—8. PMID 10393911.
- ↑ а б Hayashi G, Hagihara M, Nakatani K (2005). Application of L-DNA as a molecular tag. Nucleic Acids Symp Ser (Oxf). 49: 261—262. PMID 17150733. Помилка цитування: Некоректний тег
<ref>; назва «Hayashi2005» визначена кілька разів з різним вмістом - ↑ Vargason JM, Eichman BF, Ho PS (2000). The extended and eccentric E-DNA structure induced by cytosine methylation or bromination. Nature Structural Biology. 7: 758—761. PMID 10966645.
- ↑ Wang G, Vasquez KM (2006). Non-B DNA structure-induced genetic instability. Mutat Res. 598 (1–2): 103—119. PMID 16516932.
- ↑ Allemand та ін. (1998). Stretched and overwound DNA forms a Pauling-like structure with exposed bases. PNAS. 24: 14152—14157. PMID 9826669.
{{cite journal}}: Явне використання «та ін.» у:|author=(довідка) - ↑ Помилка цитування: Неправильний виклик тегу
<ref>: для виносок під назвоюGhoshне вказано текст - ↑ Palecek E (1991). Local supercoil-stabilized DNA structures. Critical Reviews in Biochemistry and Molecular Biology. 26 (2): 151—226. PMID 1914495.
- ↑ Basu H, Feuerstein B, Zarling D, Shafer R, Marton L (1988). Recognition of Z-RNA and Z-DNA determinants by polyamines in solution: experimental and theoretical studies. J Biomol Struct Dyn. 6 (2): 299—309. PMID 2482766.
- ↑ Leslie AG, Arnott S, Chandrasekaran R, Ratliff RL (1980). Polymorphism of DNA double helices. J. Mol. Biol. 143 (1): 49—72. PMID 7441761.
- ↑ Wahl M, Sundaralingam M (1997). Crystal structures of A-DNA duplexes. Biopolymers. 44 (1): 45—63. PMID 9097733.
- ↑ Lu XJ, Shakked Z, Olson WK (2000). A-form conformational motifs in ligand-bound DNA structures. J. Mol. Biol. 300 (4): 819—40. PMID 10891271.
- ↑ Rothenburg S, Koch-Nolte F, Haag F. DNA methylation and Z-DNA formation as mediators of quantitative differences in the expression of alleles. Immunol Rev. 184: 286—98. PMID 12086319.
- ↑ Oh D, Kim Y, Rich A (2002). Z-DNA-binding proteins can act as potent effectors of gene expression in vivo. Proc. Natl. Acad. Sci. U.S.A. 99 (26): 16666—71. PMID 12486233.
- ↑ Benham C, Mielke S (2005). DNA mechanics. Annu Rev Biomed Eng. 7: 21—53. PMID 16004565.
- ↑ Champoux J (2001). DNA topoisomerases: structure, function, and mechanism. Annu Rev Biochem. 70: 369—413. PMID 11395412.
- ↑ Wang J (2002). Cellular roles of DNA topoisomerases: a molecular perspective. Nat Rev Mol Cell Biol. 3 (6): 430—40. PMID 12042765.
- ↑ Зроблено за даними NDB UD0017
- ↑ Greider C, Blackburn E (1985). Identification of а specific telomere terminal transferase activity in Tetrahymena extracts. Cell. 43 (2 Pt 1): 405—13. PMID 3907856.
- ↑ а б Nugent C, Lundblad V (1998). The telomerase reverse transcriptase: components and regulation. Genes Dev. 12 (8): 1073—85. PMID 9553037.
- ↑ {{cite journal |author=Wright W, Tesmer V, Huffman K, Levene S, Shay J |title=Normal human chromosomes have long G-rich telomeric overhangs at one end | url=http://genesdev.org/cgi/content/full/11/21/2801 | journal=Genes Dev |volume=11 |issue=21 | pages=2801–9 |year=1997 |pmid=9353250}, що повторюються}
- ↑ а б Burge S, Parkinson G, Hazel P, Todd A, Neidle S (2006). Quadruplex DNA: sequence, topology and structure. Nucleic Acids Res. 34 (19): 5402—15. PMID 17012276.
- ↑ Griffith J, Comeau L, Rosenfield S, Stansel R, Bianchi A, Moss H, de Lange T (1999). Mammalian telomeres end in а large duplex loop. Cell. 97 (4): 503—14. PMID 10338214.
- ↑ Bird A (2002). DNA methylation patterns and epigenetic memory. Genes Dev. 16 (1): 6—21. PMID 11782440.
- ↑ Klose R, Bird A (2006). Genomic DNA methylation: the mark and its mediators. Trends Biochem Sci. 31 (2): 89—97. PMID 16403636.
- ↑ Walsh C, Xu G. Cytosine methylation and DNA repair. Curr Top Microbiol Immunol. 301: 283—315. PMID 16570853.
- ↑ Gommers-Ampt J, Van Leeuwen F, de Beer A, Vliegenthart J, Dizdaroglu M, Kowalak J, Crain P, Borst P (1993). beta-D-glucosyl-hydroxymethyluracil: а novel modified base present in the DNA of the parasitic protozoan T. brucei. Cell. 75 (6): 1129—36. PMID 8261512.
- ↑ Зроблено за даними PDB 1JDG
- ↑ Douki T, Reynaud-Angelin A, Cadet J, Sage E (2003). Bipyrimidine photoproducts rather than oxidative lesions are the main type of DNA damage involved in the genotoxic effect of solar UVA radiation. Biochemistry. 42 (30): 9221—6. PMID 12885257.
- ↑ Cadet J, Delatour T, Douki T, Gasparutto D, Pouget J, Ravanat J, Sauvaigo S (1999). Hydroxyl radicals and DNA base damage. Mutat Res. 424 (1 – 2): 9—21. PMID 10064846.
- ↑ Shigenaga M, Gimeno C, Ames B (1989). Urinary 8-hydroxy-2′-deoxyguanosine as a biological marker of in vivo oxidative DNA damage. Proc Natl Acad Sci U S A. 86 (24): 9697—701. PMID 2602371.
- ↑ Cathcart R, Schwiers E, Saul R, Ames B (1984). Thymine glycol and thymidine glycol in human and rat urine: a possible assay for oxidative DNA damage (PDF). Proc Natl Acad Sci U S A. 81 (18): 5633—7. PMID 6592579.
- ↑ Ferguson L, Denny W (1991). The genetic toxicology of acridines. Mutat Res. 258 (2): 123—60. PMID 1881402.
- ↑ Jeffrey A (1985). DNA modification by chemical carcinogens. Pharmacol Ther. 28 (2): 237—72. PMID 3936066.
- ↑ Stephens T, Bunde C, Fillmore B (2000). Mechanism of action in thalidomide teratogenesis. Biochem Pharmacol. 59 (12): 1489—99. PMID 10799645.
- ↑ Braña M, Cacho M, Gradillas A, de Pascual-Teresa B, Ramos A (2001). Intercalators as anticancer drugs. Curr Pharm Des. 7 (17): 1745—80. PMID 11562309.
- ↑ Thanbichler M, Wang S, Shapiro L (2005). The bacterial nucleoid: а highly organized and dynamic structure. J Cell Biochem. 96 (3): 506—21. PMID 15988757.
- ↑ Wolfsberg T, McEntyre J, Schuler G (2001). Guide to the draft human genome. Nature. 409 (6822): 824—6. PMID 11236998.
- ↑ Gregory T (2005). The C-value enigma in plants and animals: а review of parallels and an appeal for partnership. Ann Bot (Lond). 95 (1): 133—46.
- ↑ Pidoux A, Allshire R (2005). The role of heterochromatin in centromere function (PDF). Philos Trans R Soc Lond B Biol Sci. 360 (1455): 569—79. PMID 15905142.
- ↑ Harrison P, Hegyi H, Balasubramanian S, Luscombe N, Bertone P, Echols N, Johnson T, Gerstein M (2002). Molecular fossils in the human genome: identification and analysis of the pseudogenes in chromosomes 21 and 22. Genome Res. 12 (2): 272—80. PMID 11827946.
- ↑ Harrison P, Gerstein M (2002). Studying genomes through the aeons: protein families, pseudogenes and proteome evolution. J Mol Biol. 318 (5): 1155—74. PMID 12083509.
- ↑ Soller M (2006). Molecular fossils in the human genome: identification and analysis of the pseudogenes in chromosomes 21 and 22. Cell Mol Life Sci. 63 (7-9): 796—819. PMID 16465448.
- ↑ Michalak P. (2006). RNA world - the dark matter of evolutionary genomics. 19 (6): 1768—74. PMID 17040373.
- ↑ Cheng J, Kapranov P, Drenkow J, Dike S, Brubaker S та ін. (2005). RNA world - the dark matter of evolutionary genomics. 308: 1149—54. PMID 15790807.
{{cite journal}}: Явне використання «та ін.» у:|author=(довідка) - ↑ Mattick JS (2004). RNA regulation: а new genetics?. Nat Rev Genet. 5: 316—323. PMID 15131654.
- ↑ Albà M (2001). Replicative DNA polymerases. Genome Biol. 2 (1): REVIEWS3002. PMID 11178285.